СРАВНЕНИЕ СУБСТРАТНОЙ СПЕЦИФИЧНОСТИ РЕКОМБИНАНТНОЙ САЙТ-СПЕЦИФИЧЕСКОЙ МЕТИЛЗАВИСИМОЙ ДНК-ЭНДОНУКЛЕАЗЫ GlaI И НАТИВНОГО ФЕРМЕНТА, ВЫДЕЛЕННОГО ИЗ ШТАММА Glacial ice bacterium
Чернухин В.А., Дедков В.С., Гончар Д.А., Абдурашитов М.А., Акишев А.Г. , Наякшина Т.Н., Ломаковская Е.Н., Дегтярев С.Х.
В.А. Чернухин, В.С. Дедков, Д.А. Гончар, М.А. Абдурашитов, А.Г. Акишев, Т.Н. Наякшина, Е.Н. Ломаковская, С.Х. Дегтярев*
ООО «СибЭнзайм», 630117, Новосибирск, Россия
В настоящей работе описано клонирование сайт-специфической метилзависимой ДНК-эндонуклеазы GlaI и проведено сравнение субстратной специфичности рекомбинантного и нативного ферментов. Проведённый анализ свойств рекомбинантной эндонуклеазы GlaI показал, что субстратная специфичность нативного и рекомбинантного фермента не отличаются, но, ввиду большего количества фермента в рекомбинантном штамме, его концентрация в конечном препарате более чем в 10 раз превышает концентрацию нативного фермента.
* Автор для переписки. E-mail: degt@sibenzyme.com
Принятые сокращения: 5mC – 5-метилцитозин, MD-эндонуклеаза – метилзависимая сайт-специфическая ДНК-эндонуклеаза, ДТТ – дитиотреитол, ПААГ – полиакриламидный гель, п.н. – пары нуклеотидов, Трис – трис-(оксиметил)-аминометан, ЭДТА – этилендиаминтетрауксусная кислота, BSA – бычий сывороточный альбумин, DMSO – диметилсульфоксид, IPTG – Изопропил-бета-D-тиогалактопиранозид.
ВВЕДЕНИЕ
Метилзависимыми сайт-специфическими ДНК-эндонуклеазами, или MD-эндонуклеазами (MD – methyl dependent (англ.)) являются ферменты, которые узнают и расщепляют определенные метилированные последовательности ДНК и не гидролизуют немодифицированную ДНК [1]. По своим свойствам эти ферменты похожи на хорошо изученные эндонуклеазы рестрикции, в качестве кофакторов им необходимы только ионы магния. Фермент GlaI, выделенный из бактериального штамма Glacial ice bacterium, является MD-эндонуклеазой, которая узнает последовательность ДНК 5’-R(5mC)GY-3’ (где R – пурин, Y – пиримидин) при наличии 5-метилцитозина в комплементарной цепи ДНК, и гидролизует обе цепи ДНК посередине сайта с образованием тупых концов [2]. ДНК-метилтрансфераза DNMT3 млекопитающих и человека метилирует ДНК de novo с образованием именно последовательности 5’-R(5mC)GY-3’ [3], ввиду чего фермент GlaI нашел применение в эпигенетической ДНК-диагностике и используется для определения статуса метилирования отдельных сайтов или фрагментов ДНК [4,5], а также для установления эпигенома человека [6]. При использовании GlaI в эпигенетической ДНК-диагностике должен происходить полный гидролиз анализируемых образцов ДНК, для чего необходима высокая концентрация фермента. Однако, при выделении фермента из штамма Glacial ice bacterium для получения высокоактивного препарата фермента требуется большое количество биомассы клеток. Клонирование гена GlaI в E.coli позволяет существенно повысить содержание фермента в биомассе и получить высокоактивный рекомбинантный фермент из сравнительно небольшого количества клеток. Метилзависимые сайт-специфические ДНК-эндонуклеазы являются новыми и малоизученными ферментами, в связи с чем, мы провели сравнение субстратной специфичности нативного и рекомбинантного ферментов.
МАТЕРИАЛЫ И МЕТОДЫ
В работе использовались реактивы производства “Sigma-Aldrich” (США), “Fisher” (США), “Panreac” (Испания) и “Хеликон” (Россия). Для хроматографической очистки фермента применялись следующие носители: фосфоцеллюлоза Р-11 («Whatman», Англия), гепарин-сефароза («Bio-Rad», США), гидроксиапатит (“BioRad”, США). Наработка биомассы штамма-продуцента осуществлялась c использованием компонентов питательной среды производства ”Organotechnie” (Франция).
Штамм Esherichia coli ER2267 (F’ proA+B+ lacIq Δ(lacZ)M15 zzf::mini-Tn10 (Kanr)/Δ(argF-lacZ)U169 glnV44 e14- (McrA-) rfbD1. recA1 relA1. endA1 spoT1. Thi-1 Δ(mcrC-mrr) 114::IS10) получен из “New England Biolabs, Inc.” (США). Для экспериментов использовали препараты ДНК фага λ и различных С5-метилированных плазмид, которые были сконструированы ранее [7-9].
В качестве маркёра длины фрагментов ДНК при получении электрофореграмм в агарозных гелях использовали ДНК-маркёры 1 kb («СибЭнзим», Россия). Для разбавления препарата рекомбинатного GlaI использовался SE-буфер для хранения и разбавления ферментов «B100» (10 мМ Трис-HCl (pH 7.6), 50 мМ KCl, 0.1 мМ ЭДТА, 200 мкг/мл BSA, 1 мМ ДТТ, 50% глицерин) («СибЭнзим», Россия). Все экспериметы по анализу активности и субстратной специфичности рекомбинантного и нативного GlaI проводились в 20 мкл реакционной смеси, содержащей SE-буфер «Y» (33 мМ Трис-ацетат (pH 7.9), 10 мМ магния ацетат, 66 мМ калия ацетат, 1 мМ ДТТ) и 0,5 мкг субстратной ДНК при оптимальной температуре 30°С в течение 1 часа.
Клонирование GlaI.
Бактериальная ДНК штамма Glacial ice bacterium была выделена как описано ранее [10] и гидролизовалась эндонуклеазой рестрикции Kzo9I. Фрагменты ДНК клонировались в вектор pUC19 [11], линеаризованный BamHI, и далее по стандартной методике [12] была получена библиотека рекомбинантных клонов E.coli ER2267, несущих плазмиды со встроенными фрагментами ДНК Glacial ice bacterium. Полученные клоны анализировались на предмет наличия активности GlaI в лизате с использованием в качестве субстрата ДНК плазмиды pHspAI [9], гидролизованной рестриктазой DriI (pHspAI/DriI), как описано ранее [13].
Наработка биомассы рекомбинантного штамма E.coli ER2267 (pMTL22-GlaI), несущего плазмиду с геном MD-эндонуклеазы GlaI.
Для получения биомассы одиночную колонию штамма E.coli ER2267 (pMTL22-GlaI) засевали во флакон со 100 мл питательной среды LB (1% триптон, 0.5% дрожжевой экстракт, 0.5% NaCl, рН 7.5) с ампициллином (50 мкг/мл). Культуру подращивали в течение ночи в термостате при 37°С. Затем полученный инокулят засевали по 5 мл в 12 флаконов со 100 мл среды LB с ампициллином. Культуру подращивали в течение 16 часов в термостатированной качалке при 30°С с перемешиванием при 140 об/мин. После 11 часов выращивания в культуру добавляли IPTG до концентрации 1 мМ и растили еще в течение 5 часов. Затем 1 мл культуральной жидкости отбирался для анализа, а остальная культура центрифугировалась (J2-21 «Beckman», США) при 6000 об/мин в течение 30 мин. Осадок клеток замораживали при -20°С.
Для анализа целевой активности 1 мл культуральной жидкости откручивали на центрифуге Eppendorf при 13000 об/мин в течение 2 мин, и полученный осадок клеток суспендировали в 100 мкл лизирующего буфера (10 мМ Трис-HCl, pH 8.5; 0.1 мг/мл лизоцима, 50 мМ NaCl, 1 мМ ЭДТА, 0,1% Тритон Х-100) и встряхивали в течение 30 мин на «Multi-vortex V-32» («Biosan», Литва). 8 мкл полученного лизата клеточной культуры добавляли к 40 мкл реакционной смеси, содержащей 1х SE-буфер «Y» и 0,5 мкг ДНК плазмиды pНspAI/DriI, использованной ранее для тестирования GlaI из дикого штамма [9]. Смесь пипетировали, после чего 20 мкл этой смеси переносили в следующую лунку с 20 мкл той же реакционной смеси, в результате чего получали реакционную смесь, содержащую в 2 раза меньше лизата. Аналогичной процедурой получали всего 8 последовательных разведений лизата в реакционной смеси с SE-буфером «Y» и ДНК pНspAI/DriI. Смесь инкубировали в течение 1 часа при температуре 30°С, после чего реакцию останавливали стоп-буфером (40% сахароза, 0.1 М ЭДТА, 0.05% бромфеноловый синий).
Выделение препарата фермента GlaI из биомассы.
Выделение фермента проводили при 4°C путем колоночной хроматографии. 10 г биомассы штамма E.coli ER2267 (pMTL22-GlaI) суспендировали в 30 мл буфера А (10 мМ Tris-HCl, pH 7.5, 0.1 мМ ЭДТА, 7 мМ β-меркаптоэтанол), содержащего 0.3 М NaCl, 0.1 мг/мл лизоцим, 1 мМ фенилметилсульфонилфторид (PMSF) и 0.1% Тритон X-100. Биомассу лизировали в стеклянном стакане при перемешивании на магнитной мешалке в течение 1 часа. Полученный лизат клеток обрабатывали ультразвуком на дезинтеграторе Soniprep 150 («MSE», Англия) с диаметром адаптера 2 см. Дезинтеграцию ультразвуком проводили при амплитуде 20 мкм 5 раз по 30 секунд с интервалами в 1 мин с охлаждением суспензии в ледяной бане.
Далее лизат осветляли центрифугированием на центрифуге J2-21 («Beckman», США) при 15000 об/мин в течение 30 мин. После чего супернатант наносили на колонку с фосфоцеллюлозой Р-11 объёмом 20 мл, предварительно уравновешенную буфером А, содержащим 0.1 М NaCl. Колонку промывали 40 мл буфера А с 0.1 М NaCl, затем проводили элюцию целевого белка линейным градиентом NaCl (0.1-0.65 М) в буфере А объёмом 200 мл, собирая фракции по 4 мл. Фракции, содержащие MD-эндонуклеазу, объединяли, диализовали против 20 объёмов буфера А с 0.1 М NaCl и наносили на колонку с 10 мл гепарин-сефарозы. Элюцию фермента проводили линейным градиентом NaCl (0.1-0.45 М) в буфере А объёмом 200 мл, собирая фракции по 4 мл. Целевые фракции объединяли и наносили на колонку с гидроксиапатитом объемом 4 мл, предварительно промытую уравновешивающим буфером Б (10 мМ K-фосфат, pH 7.2, 0.1 мM ЭДТА, 7 мМ β-меркаптоэтанол, 0.05 M NaCl). Колонку промывали 10 мл буфера Б и проводили элюцию фермента линейным градиентом K-фосфата (0.01-0.4 М) в буфере Б объемом 100 мл, собирая фракции по 2.5 мл. Фракции, содержащие целевой белок, объединяли и диализовали против 20 объёмов концентрирующего буфера (10 мМ Трис-HCl, pH 7.5, 0.1 М NaCl, 0.1 мМ ЭДТА, 0.05% Тритон Х-100, 7 мМ β-меркаптоэтанол, 50% глицерин). Полученный препарат фермента GlaI хранили при -20°C.
РЕЗУЛЬТАТЫ И ОБСУЖДЕНИЕ
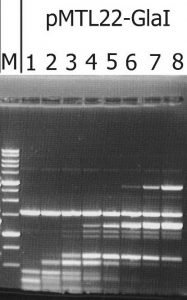
По результатам определения активности MD-эндонуклеазы GlaI в лизатах клонов библиотеки pUC19/BamHI-Gla DNA/Kzo9I с использованием ДНК-субстрата pHspAI/DriI был выделен рекомбинантный штамм E.coli, несущий плазмиду с целевым геном glaI, который затем был переклонирован в плазмидный вектор pMTL22 [14] по сайтам рестрикции FauNDI и BamHI. Сконструированной плазмидой pMTL22-GlaI были трансформированы клетки E.coli ER2267 и получен рекомбинантный штамм E.coli ER2267 (pMTL22-GlaI), который далее использовался для наработки биомассы и последующего выделения фермента. Результаты тестирования целевой активности в лизате клеток полученной биомассы рекомбинантного штамма E.coli ER2267 (pMTL22-GlaI) приведены на рисунке 1.
Рисунок 1. Анализ активности целевого фермента в биомассе рекомбинантного штамма E.coli ER2267 (pMTL22-GlaI) – продуцента GlaI, на ДНК плазмиды pHspAI/DriI. Электрофорез в 1% агарозном геле. Дорожки: 1-8 – добавлены разведения лизата биомассы клеток как описано в «Материалах и Методах»; М – ДНК-маркёры 1kb
Как видно из рисунка, на дорожке 1 наблюдается полный гидролиз субстратной ДНК, и картина гидролиза соответствует набору фрагментов, характерному для нативного фермента GlaI [9].
В результате выделения MD-эндонуклеазы GlaI путем хроматографической очистки из 10 г клеток рекомбинантного штамма-продуцента было получено 6 мл препарата фермента с активностью 120,000 е.а./мл, что более чем в 10 раз превышает активность препаратов, получаемых из дикого штамма-продуцента GlaI (до 10,000 е.а./мл).
Анализ субстратной специфичности рекомбинантного GlaI.
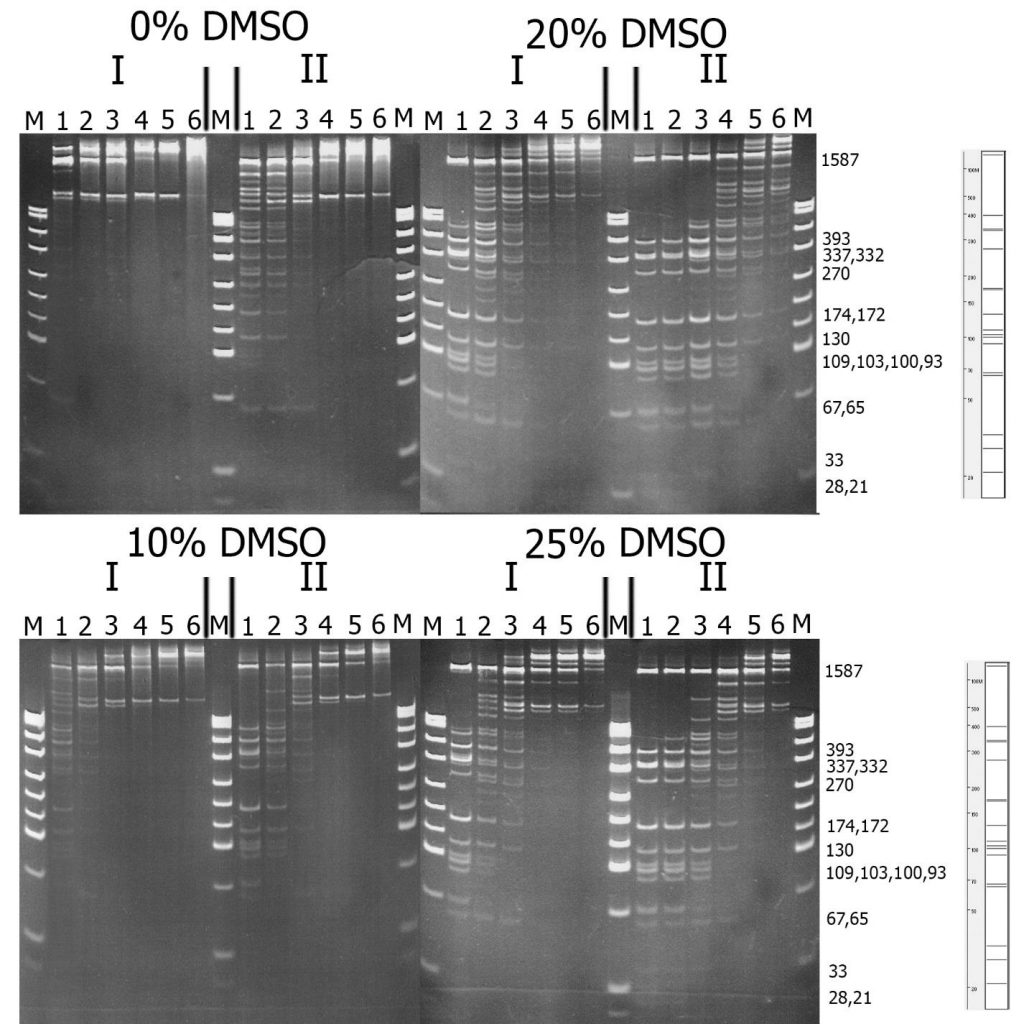
Ранее было установлено, что GlaI, выделенный из дикого штамма, имеет в 20-30 раз большую активность в присутствии 20% DMSO [15]. Поэтому нами было проведено сравнение субстратной специфичности рекомбинантного и нативного GlaI при гидролизе ДНК pHspAI/DriI в присутствии разных концентраций DMSO. Реакции гидролиза проводились в SE-буфере «Y» в отсутствие или присутствии DMSO (10%, 20%, 25%) при оптимальной температуре (30°C) в течение 1 часа.
На рисунке 2 представлено сравнение активностей препаратов GlaI, выделенных из дикого и рекомбинантного штаммов. Для удобства сравнения препарат рекомбинантного GlaI был предварительно разбавлен буфером для разбавления ферментов «В100» в 4 раза.
Рисунок 2. Сравнение субстратной специфичности фермента GlaI, выделенного из дикого (I) и рекомбинантного (II) штамма, на ДНК pHspAI/DriI. Электрофорез в 12% ПААГ. Справа приведена теоретически рассчитанная картина гидролиза плазмиды pHspAI/DriI по сайтам 5’-R(5mC)GY-3’ (крайняя справа) и указаны длины получаемых фрагментов ДНК. Дорожки: 1-6 – последовательные разведения препарата фермента в 2 раза, М – ДНК-маркёры pUC19/MspI.
Из данного рисунка видно, что рекомбинантный фермент GlaI, подобно нативному ферменту, активируется присутствием в реакционной смеси DMSO, причём наибольшая активность проявляется при концентрации DMSO 20%. Из рисунка видно также, что субстратная специфичность обоих препаратов на pHspAI/DriI не отличается, однако обработка ДНК рекомбинантным препаратом фермента, даже разбавленным в 4 раза, приводит к более полному гидролизу субстрата по сравнению с нативным препаратом. Картина гидролиза ДНК на дорожке 1 (нативный фермент), чуть лучше, чем на дорожке 3 (рекомбинантный фермент), что соответствует более чем 10-тикратному превышению концентрации клонированного GlaI.
Далее мы проводили сравнительный анализ активности нативного и рекомбинантного ферментов на различных 5-метилцитозин содержащих ДНК. В качестве контроля использовали ДНК фага λ. В работе использовали следующие плазмиды:
- pMHpaII (несёт ген метилазы M.HpaII, метилирующей второй цитозин в последовательности 5′-CСGG-3′, и содержит метилированные сайты 5’-C(5mC)GG-3’/3’-GG(5mC)C-5’ [16]);
- pMHaeIII (несёт ген метилазы M.HaeIII, метилирующей первый цитозин в последовательности 5′-GGCС-3′, и содержит метилированные сайты 5’- GG(5mC)C -3’/3’- C (5mC) GG -5’ [17]);
- pFsp4HI3 (несёт ген метилазы M.Fsp4HI, метилирующей первый цитозин в последовательности 5′-GCNGC-3′, и содержит метилированные сайты 5’-G(5mC)NGC-3’/3’-CGN(5mC)G-5’ [8]);
- pBspACI (несёт гены метилаз M1.BspACI и M2.BspACI и содержит метилированные последовательности 5’- (5mC)CGC -3’/3’- GG(5mC)G -5’ [18]);
- pHspAI (несёт ген метилазы M. HspAI, метилирующей первый цитозин в последовательности 5′-GCGC-3′, и содержит метилированные сайты 5’-G(5mC)GC-3’/3’-CG(5mC)G-5’ [9]).
Поскольку гидролиз ДНК удобнее анализировать не на кольцевой, а на линейной форме ДНК, все плазмиды были предварительно линеаризованы с помощью эндонуклеазы рестрикции DriI. Реакции проводились в SE-буфере «Y» в присутствии 20% DMSO.
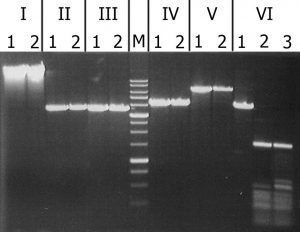
На рис. 3 представлена электрофореграмма продуктов расщепления данных субстратов рекомбинантным GlaI.
Рисунок 3. Анализ субстратной специфичности рекомбинантного GlaI. Электрофорез в 1% агарозном геле. Дорожки: I – ДНК фага λ; II – pMHpaII/DriI; III – pMHaeIII/DriI; IV- pFspAI3/DriI; V – pBspACI/DriI; VI – pHspAI/DriI. 1 – ДНК без добавления фермента; 2 – ДНК с добавлением рекомбинантного GlaI; 3 – ДНК с добавлением нативного GlaI (контроль); М – ДНК-маркёры 1kb.
Из рисунка видно, рекомбинантный GlaI не расщепляет неметилированный субстрат (ДНК фага λ), а также все С5-метилированные субстраты, кроме плазмиды pHspAI. Из представленных данных можно заключить, что субстратная специфичность рекомбинантного и нативного фермента совпадают. Фермент GlaI узнаёт и расщепляет последовательность 5’-G(5mC)GC-3’/3’-CG(5mC)G-5’ и не гидролизует другие сайты с 5-метилцитозином в обеих цепях ДНК [2].
Полученные результаты подтверждают, что препарат фермента GlaI, полученный из рекомбинантного штамма, обладая на порядок большей активностью, проявляет при этом ту же субстратную специфичность, что и фермент, выделенный из исходного штамма Glacial ice bacterium. Таким образом, использование рекомбинантного GlaI позволяет добиться более полного гидролиза анализируемых образцов ДНК, что несомненно улучшит результаты эпигенетических исследований с применением этого фермента.
ЛИТЕРАТУРА
- Methyl-directed DNA endonucleases
- Тарасова Г.В., Наякшина Т.Н., Дегтярев С.Х. Субстратная специфичность новой ДНК-эндонуклеазы GlaI. // BMC Molecular Biology. 2008. 9:7.
- Handa V., Jeltsch A. Profound flanking sequence preference of Dnmt3a and Dnmt3b mammalian DNA methyltransferases shape the human epigenome. // J. Mol. Biol. 2005. 348(5): 1103-1112.
- Gonchar D.A., Akishev A.G., Degtyarev S.Kh. BlsI- and GlaI-PCR assays – a new method of DNA methylation study. // Ovchinnikov bulletin of biotechnology and physical and chemical biology. 2010. V.6. No. 1. P. 5-12.
- Kuznetsov V.V., Abdurashitov M.A., Akishev A.G., Degtyarev S.H. Method of determining nucleotide sequence Pu(5mC)GPy at predetermined position of long-distance DNA. // Russian Federation patent RU 2525710 C1. 2014.
- Abdurashitov M.A., Tomilov V.N., Gonchar D.A., Kuznetsov V.V., Degtyarev S.Kh. Mapping of R(5mC)GY Sites in the Genome of Human Malignant Cell Line Raji. // Biol. Med. (Aligarh). 2015. V. 7. Is. 4. BM-135-15.
- Чмуж Е.В., Каширина Ю.Г., Томилова Ю.Э., Мезенцева Н.В., Дедков В.С., Гончар Д.А., Абдурашитов М.А., Дегтярев С.Х. Новая эндонуклеаза рестрикции BisI из Bacillus subtilis Т30 узнает метилированную последовательность ДНК 5’G(5mC)^NGC-3’. // Биотехнология. 2005. №3. С.22-26.
- Чернухин В.А., Чмуж Е.В., Томилова Ю.Э., Наякшина Т.Н., Гончар Д.А., Дедков В.С., Дегтярев С.Х. Новая сайт-специфичесчкая эндонклеаза GluI узнаёт метилированную последовательность ДНК 5’-G(5mC)NG(5mC)-3’/3’-(5mC)GN(5mC)G-5’. // Вестник биотехнологии и физико-химической биологии имени Ю.А. Овчинникова. 2007. Т.3. №2. С.13-17.
- Чернухин В.А., Наякшина Т.Н., Абдурашитов М.А., Томилова Ю.Э., Мезенцева Н.В., Дедков В.С., Михненкова Н.А., Гончар Д.А., Дегтярев С.Х. Новая эндонуклеаза рестрикции GlaI узнаёт метилированную последовательность 5’-GCGC-3’. // Биотехнология. 2006. №4. С.31–35.
- Smith C.L., Klco S.R. and Cantor C.R. In: Davies K. (ed.). Genome Analysis: A Practical Approach. 1987. IRL Press. Oxford. UK.
- Yanisch-Perron C., Vieira J., Messing J. Improved M13 phage cloning vectors and host strains: nucleotide sequences of the M13mp18 and pUC19 vectors. // Gene. 1985. V.33. P.103-119.
- Sambrook J., Fritsch E.F., Maniatis T. Molecular cloning: a laboratopy manual. 1987. Cold Spring Harbor Laboratory Press, USA.
- Дедков В.С., Гончар Д.А., Чернухин В.А., Абдурашитов М.А., Ломаковская Е.Н., Шинкаренко Н.М., Дегтярёв С.Х. Клонирование системы рестрикции-модификации BpuN4I из Bacillus pumilus N4. // Вестник биотехнологии и физико-химической биологии имени Ю. А. Овчинникова. 2013. Т.9. №3. С.40-47.
- Chambers S.P., Prior S.E., Barstow D.A., Minton N.P. The pMTLnic— cloning vectors. I. Improved pUC polylinker regions to facilitate the use of sonicated DNA for nucleotide sequencing. // Gene. 1998. V.68. P.139-149.
- Abdurashitov M.A., Chernukhin V.A., Gonchar D.A., Dedkov V.S., Mikhnenkova N.A. and Degtyarev S.Kh. Dimethyl Sulfoxide Changes the Recognition Site Preference of Methyl- directed Site-specific DNA Endonuclease GlaI. // Research Journal of Pharmaceutical, Biological and Chemical Sciences. 2016. №7(1). P.1733-1739.
- Чернухин В.А., Килева Е.В., Томилова Ю.Э., Болтенгаген А.А., Дедков В.С., Михненкова Н.А., Гончар Д.А., Голикова Л.Н., Дегтярев С.Х. Новая метилзависимая сайт-специфическая эндонуклеаза KroI узнает и расщепляет последовательность ДНК 5`-G^C(5mC)GGC-3`. // Вестник биотехнологии и физико-химической биологии имени Ю. А. Овчинникова. 2011. Т.7. №1. С.14-20.
- Чернухин В.А., Беличенко О.А., Тарасова Г.В., Гончар Д.А., Акишев А.Г., Дедков В.С., Михненкова Н.А., Дегтярев С.Х. Штамм бактерии Arthrobacter oxydans — продуцент сайт-специфической эндонуклеазы AoxI. // Патент на изобретение RU 2399663 С1. 2009.
- Тарасова М.В., Джанобилова З.К., Томилова Ю.Э., Дедков В.С., Чернухин В.А., Дегтярев С.Х. Новая сайт-специфическая эндонуклеаза BspACI из BACILLUS PSYCHRODURANS AC узнает последовательность 5`-C^CGC-3`/3`-GGC^G-5`. // Вестник биотехнологии и физико-химической биологии имени Ю.А.Овчинникова. 2009. Т.5. №1. С.16-24.