Выщепление рестриктазой амплифицируемого фрагмента ДНК как способ исключения ошибочных результатов ПЦР в реальном времени c TaqMan зондом
Акишев А.Г., Нетесова Н.А., Абдурашитов М.А., Дегтярев С.Х.
А.Г. Акишев1 *, Н.А. Нетесова2, М.А. Абдурашитов1, С.Х. Дегтярев1
1 ОOO «СибЭнзайм» Новосибирск
2 ООО «Эпигенлаб» Новосибирск
* автор для переписки: Александр Акишев, ООО СибЭнзайм, ул. Тимакова, д. 2/12 , Новосибирск, 630117, Россия; тел.: +7(383)333-4991; факс: +7(383)333-6853; E-mail: aki@sibenzyme.ru
ПЦР в реальном времени с использованием TaqMan зонда (ПЦР РВ) является одним из основных методов анализа структуры ДНК человека и широко используется в медицине. В данной работе проводили ПЦР РВ 4-х фрагментов геномной ДНК человека (chr1: 90717178-90717300, chr9: 97464604-97464756, chr11: 65647215-65647307, chr17: 4560027-4560175), используя в качестве матрицы препараты ДНК из крови человека, полученные как стандартным методом фенольной депротеинизации, так и выделенные на колонках. В ПЦР использовались как препараты исходной ДНК, так и после ее обработки рестриктазой, выщепляющей амплифицируемый фрагмент. В случае препаратов нативной ДНК, очищенных на колонках, ПЦР на 3-х из 4-х фрагментов ДНК дает завышенное значение Cq. В случае фенольной очистки завышенное значение Cq для препаратов нативной ДНК получается для 2-х из 4-х фрагментов ДНК. В случае препаратов ДНК, полученных после очистки на колонке, обработка ДНК эндонуклеазой рестрикции, вырезающей амплифицируемый фрагмент, приводит к получению нормальных значений Cq для всех четырех анализируемых фрагментов ДНК. Таким образом, ПЦР РВ с нативной ДНК, полученной очисткой на колонках, может давать завышение Cq, которое снимается, если предварительно провести гидролиз ДНК рестриктазой, выщепляющей ампликон.
Ключевые слова: рестриктазы, препараты ДНК из крови человека, ПЦР РВ
DOI: 10.26213/SE.2021.45.29.001
Данные для цитирования:
Акишев А.Г., Нетесова Н.А., Абдурашитов М.А., Дегтярев С.Х. (2021) Выщепление рестриктазой амплифицируемого фрагмента ДНК как способ исключения ошибочных результатов ПЦР в реальном времени c TaqMan зондом, ДНК-узнающие ферменты, том 2021(1), DOI: 10.26213/SE.2021.45.29.001

Эта статья доступна по лицензии Creative Commons Attribution-NonCommercial-NoDerivatives 4.0 International
ВВЕДЕНИЕ
На сегодня ПЦР в реальном времени с использованием TaqMan зонда (ПЦР РВ) повсеместно используется при анализе структуры ДНК человека и является одним из основных диагностических методов в медицине. Ранее мы показали, что использование рестриктаз для предварительного гидролиза ДНК-матрицы с целью выщепления амплифицируемого фрагмента позволяет проводить сравнительные исследования образования ПЦР-продукта на большой выборке препаратов ДНК [1-3]. В данной работе мы проводили ПЦР РВ 4-х фрагментов геномной ДНК человека, используя в качестве матрицы препараты ДНК из крови человека, полученные как стандартным методом фенольной депротеинизации, так и выделенные на колонках. При этом, ПЦР ставилась как на исходной ДНК, так и после ее обработки рестриктазой, в результате действия которой происходит вырезание амплифицируемого фрагмента.
МАТЕРИАЛЫ И МЕТОДЫ
Материалы. В работе использовали фенол (ОАО «Реактив», Новосибирск), хлороформ (ОАО «Реактив», Новосибирск), изоамиловый спирт (ОАО «Реактив», Новосибирск), РНКазу А (ООО «Самсон-Мед», Санкт-Петербург), SDS (ООО «Компания Хеликон», Москва). Реакционная смесь для пробоподготовки ДНК перед ПЦР, набор для очистки ДНК из легкой фракции клеток крови, олигонуклеотиды, рестриктазы Bst2UI, HinfI, MspI, бидистиллированная вода и буферные растворы производства ООО «СибЭнзайм».
Материалом для исследования служили образцы ДНК, выделенные из клеток крови 16 условно здоровых доноров. Препараты ДНК 8-ми доноров под номерами 205, 1086, 1087, 1137, 1151, 1205, 1437 и 1487 получали фенол-хлороформным методом как описано ранее [4] с некоторыми модификациями. В ходе очистки полученные препараты ДНК обрабатывались только РНКазой А без добавления рестриктазы TaqI. В случае других 8-ми препаратов под номерами 232, 235, 582, 558, 559, 604, 605 и 640 выделение ДНК проводили из легкой фракции клеток крови с доочисткой на спин-колонках как описано ранее [5]. Концентрацию полученных препаратов ДНК определяли спектрофотометрически.
Приготовление рестрикционной смеси проводилось как описано ниже. При проведении ПЦР РВ фрагмента ДНК 9-604 в пробирку на 1,5 мл отбирали «реакционную смесь для пробоподготовки ДНК перед ПЦР» в объеме 250 мкл и к ней добавляли 1 мкл (20 ед.акт) эндонуклеазы рестрикции HinfI (смесь А). При проведении ПЦР РВ фрагмента ДНК 1-178 в пробирку на 1,5 мл отбирали «реакционную смесь для пробоподготовки ДНК перед ПЦР» в объеме 250 мкл и к ней добавляли 1 мкл (20 ед.акт) эндонуклеазы рестрикции Bst2UI (смесь Б). При проведении ПЦР РВ фрагментов ДНК 17-027 и 11-215 в пробирку на 1,5 мл отбирали «реакционную смесь для пробоподготовки ДНК перед ПЦР» объеме 250 мкл и к ней добавляли 1 мкл (20 ед.акт) эндонуклеазы рестрикции MspI (смесь В).
Приготовление ПЦР-смесей. Для каждого из четырех фрагментов (с учетом того, что имеется 16 гидролизованных и 16 негидролизованных образцов ДНК) готовилось 940 мкл реакционной смеси для ПЦР, содержащей 714 мкл воды, 205 мкл 10х буфера для Hot Start Taq ДНК-полимеразы и 21 мкл 10 мМ смеси дезоксинуклеозидтрифосфатов. К полученной смеси добавляли 77 мкл смеси праймеров и зонда (10 µM каждый), специфичные для исследуемого фрагмента ДНК, и 12,8 мкл Hot Start Taq ДНК-полимеразы (5 ед.акт/мкл). Полученную ПЦР-смесь перемешивали.
Гидролиз ДНК крови рестриктазами проводили в пробирках для ПЦР (200 мкл). В каждой пробирке смешивали 13,5 мкл H2O и 1,5 мкл ДНК образца, доведенного до концентрации около 18 нг/мкл. Для каждого исследуемого образца готовили по 2 пробирки ДНК. В одну пробирку добавляли 15,1 мкл реакционной смеси для пробоподготовки ДНК перед ПЦР, а в другую пробирку добавляли 15,1 мкл смеси А в случае анализа фрагмента ДНК 9-604, 15,1 мкл смеси Б в случае анализа фрагмента ДНК 1-178 или 15,1 мкл смеси В случае анализа фрагментов ДНК 17-027 и 11-215. Для фрагмента ДНК 1-178 инкубацию проводили при 60 С, в остальных случаях – при 37 С. Время инкубации 30 минут.
Проведение ПЦР. После инкубации сбрасывали капли на микроцентрифуге и в каждую пробирку добавляли по 30,2 мкл ПЦР-смеси. После перемешивания из каждой пробирки ее содержимое (60 мкл) разносили по 20 мкл в лунку ПЦР-планшетки на 96 лунок.
Для расчета специфических праймеров и зондов использовали нуклеотидные последовательности из базы данных GenBank (http://ncbi.nlm.nih.gov/genbank) по версии генома человека GRCh38/hg38, семейство программ “Vector NTI 11.5” (Invitrogen, США) и онлайн-ресурс «BLAST» (http://blast.ncbi.nlm.nih.gov). Ниже указана структура праймеров и флуоресцентно меченого зонда, используемых в работе:
9-604z 5′ FAM-TGTGAGCCACTGTGACCAGCCCCC-BHQ1 3′(24)
9-604d 5′ CCTCGGCCTCCCAGAGTGC 3′(19)
9-604r 5′ CCTGCTGCCATCTTTGGTTCC 3′ (21)
17-027z 5′ TCAAAGGTCCCCAGCCCCCAA 3′(21)
17-027r 5′ CCAAGCCAGCCTCTGTCCCA 3′(20)
17-027d 5′ GGGGTGTGGCAGGCAGGAG 3′(19)
11-215z 5′ CCACCGCCCGAGGGATGAAGACC 3′(23)
11-215d 5′ TGGTGCCCTCGGGTAAGCG 3′(19)
11-215r 5′ CCC AGG CCA CAC GGA ACT TTC 3′(21)
1-178z 5′ CCGCCGTTCAGCAGCCGCCC 3′(20)
1-178r 5′ CGGCGGCAACATTGTTTGGT 3′(20)
1-178d 5′ GGC CCC TTC CAT TGT CAT TGC T 3′(22)
ПЦР РВ проводили согласно протоколу производителя [6] в объеме 20 мкл на детектирующем амплификаторе CXF-96 («Bio-Rad», США) по следующей программе:
для фрагментов 9-604, 17-027, 1-178:
- при 95°C — 3 мин;
- 5 «слепых» циклов: 95°C — 10 сек, 69°C (с понижением на 0.7°C в каждом последующем цикле) — 20 сек, 72°C — 10 сек, 75°C — 5 сек;
- 40 циклов: 95°C — 10 сек, 65°C — 20 сек (с детекцией флуоресцентного сигнала в канале FAM), 72°C — 10 сек, 75°C — 5 сек;
для фрагмента 11-215:
- при 95°C — 3 мин;
- 5 «слепых» циклов: 95°C — 10 сек, 69°C (с понижением на 0.5°C в каждом последующем цикле) — 20 сек, 72°C — 10 сек, 75°C — 5 сек;
- 40 циклов: 95°C — 10 сек, 66°C — 20 сек (с детекцией флуоресцентного сигнала в канале FAM), 72°C — 10 сек, 75°C — 5 сек.
По завершении ПЦР, при помощи программного обеспечения амплификатора “Bio-Rad CFX Manager v.2.1”, устанавливали среднее значение Cq и значение среднеквадратичного отклонения для анализируемых образцов и полученные данные заносили в таблицу.
РЕЗУЛЬТАТЫ И ОБСУЖДЕНИЕ
В работе изучались препараты ДНК крови 16 доноров, полученные как стандартным методом фенольной очистки, так и с помощью набора для выделения ДНК [7]. В числе доноров были 7 мужчин и 9 женщин. В таблице 1 представлены пол и возраст доноров (столбики 2 и 3, соответственно), а также концентрация полученных препаратов ДНК (столбик 4) и характеристика их чистоты, определяемой отношением оптической плотности при 260 нм и 280 нм (столбик 5). Синим фоном выделены номера препаратов ДНК, полученные фенольной очисткой.
Таблица 1
Пол и возраст доноров крови, концентрация и фотометрическая чистота полученной ДНК крови.
|
N донора |
Пол |
Возраст, лет |
Концентрация ДНК, нг/мкл |
А260/А280 |
|
232 |
ж |
42 |
74 |
1,80 |
|
235 |
м |
54 |
35 |
1,90 |
|
558 |
ж |
68 |
54 |
1,90 |
|
559 |
ж |
66 |
20 |
1,90 |
|
582 |
м |
33 |
54 |
1,90 |
|
604 |
ж |
36 |
52 |
1.90 |
|
605 |
м |
54 |
50 |
2,00 |
|
640 |
м |
43 |
48 |
1,80 |
|
205 |
ж |
72 |
461 |
1.80 |
|
1086 |
м |
36 |
193 |
1.70 |
|
1087 |
ж |
51 |
95 |
1.70 |
|
1137 |
ж |
59 |
299 |
1.60 |
|
1151 |
ж |
50 |
225 |
1.70 |
|
1205 |
м |
65 |
420 |
1.70 |
|
1437 |
м |
68 |
213 |
1.80 |
|
1487 |
ж |
22 |
406 |
1.70 |
Как видно из таблицы, выделенные препараты ДНК имеют концентрацию в диапазоне от 20 нг/мкл до 461 нг/мкл и чистоту не ниже 1,6 по соотношению А260/А280.
C полученными препаратами ДНК, как исходными, так и после расщепления одной из трех эндонуклеаз рестрикции (Bst2UI, HinfI, MspI) , проводилась ПЦР в реальном времени, как указано в «Материалах и Методах». Для каждого из 4-х амплифицируемых фрагментов гидролиз препаратов ДНК рестриктазами Bst2UI (фрагмент 1-178), HinfI (фрагмент 9-604) и MspI (фрагменты 17-027 и 11-215) приводит к выщеплению анализируемого фрагмента, что позволяет избежать влияния остальной ДНК на результаты ПЦР.
Нами проводился ПЦР РВ 4-х фрагментов ДНК, название которых соответствует позиции 5’-конца ампликона в геноме и включает номер хромосомы и последние три цифры его положения. Например, название фрагмента 9-604 соответствует положению его 5′-конца в позиции chr9: 97464604.
На рисунке 1 представлены нуклеотидные последовательности 4-х участков геномной ДНК, анализируемые в данной работе. В каждом участке ДНК фиолетовым фоном выделены сайты узнавания рестриктазы, окаймляющие амплифицируемый участок. Зоны связывания прямого и обратного праймеров, а также флуоресцентно-меченого TaqMan-зонда выделены желтым, голубым и зеленым фоном, соответственно.
Рисунок 1
Нуклеотидные последовательности участков генома, выбранных для проведения ПЦР.
chr9: 97464604-97464756 – amplicon 153 bp, GC-состав 49%, гидролиз HinfI |
|
|
chr17: 4560027-4560175 – amplicon 149 bp, GC-состав 67%, гидролиз MspI |
|
|
chr 11: 65647215-65647307 – amplicon 93 bp, GC-состав 68%, гидролиз MspI |
|
|
chr 1: 90717178-90717300 – amplicon 123 bp, GC-состав 54%, гидролиз Bst2UI |
|
|
На рисунке 2 представлены кривые флуоресценции ПЦР РВ для образцов ДНК №1151 (выделение фенольным методом) и №559 (выделение на колонке).
Рисунок 2
Кривые флуоресценции ПЦР-анализа 4-х фрагментов генома человека в препаратах ДНК доноров №559 и №1151.
9-604 |
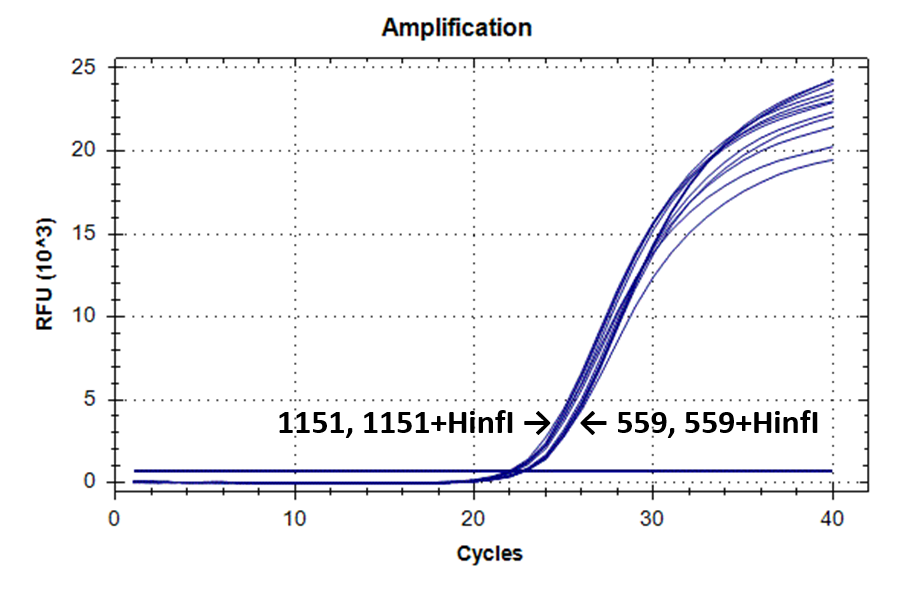 |
17-027 |
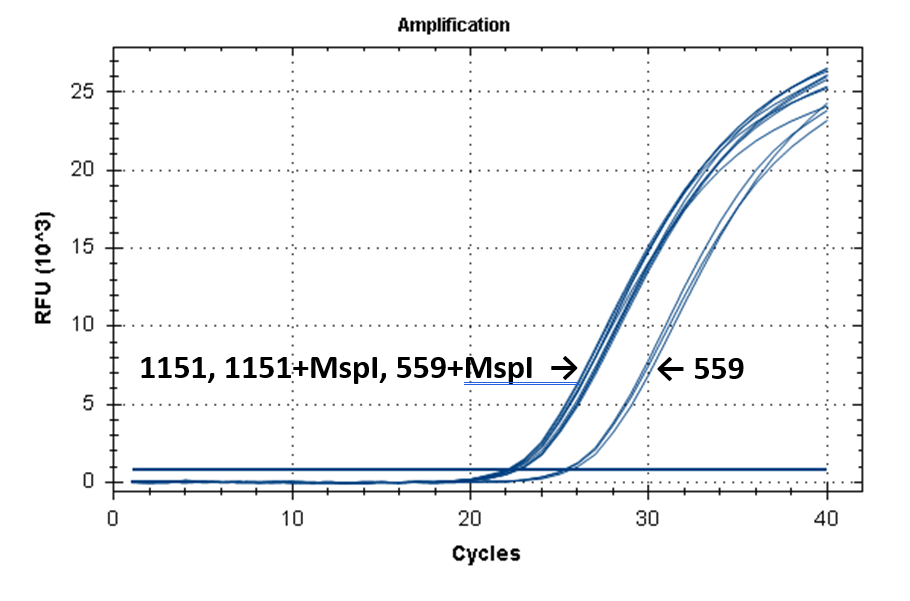 |
11-215 |
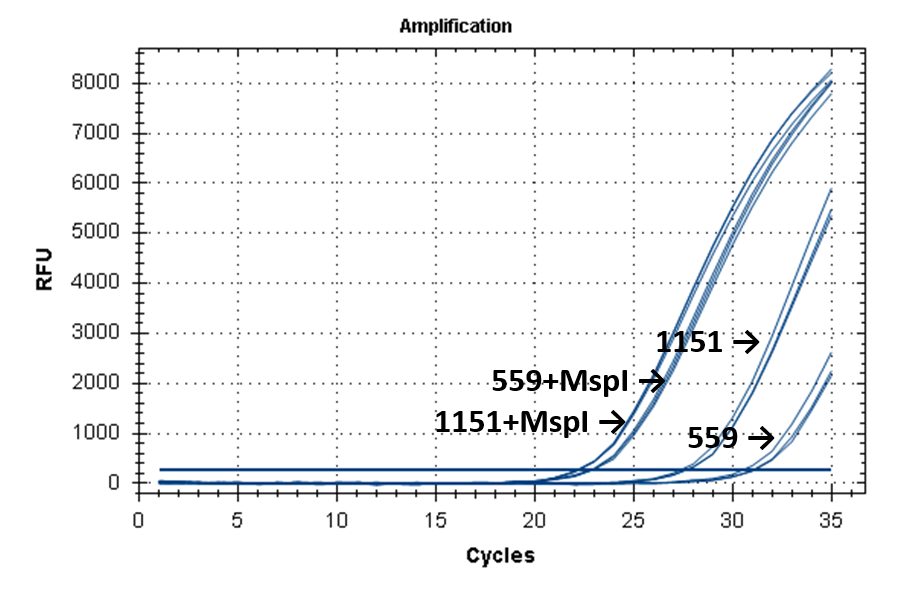 |
1-178 |
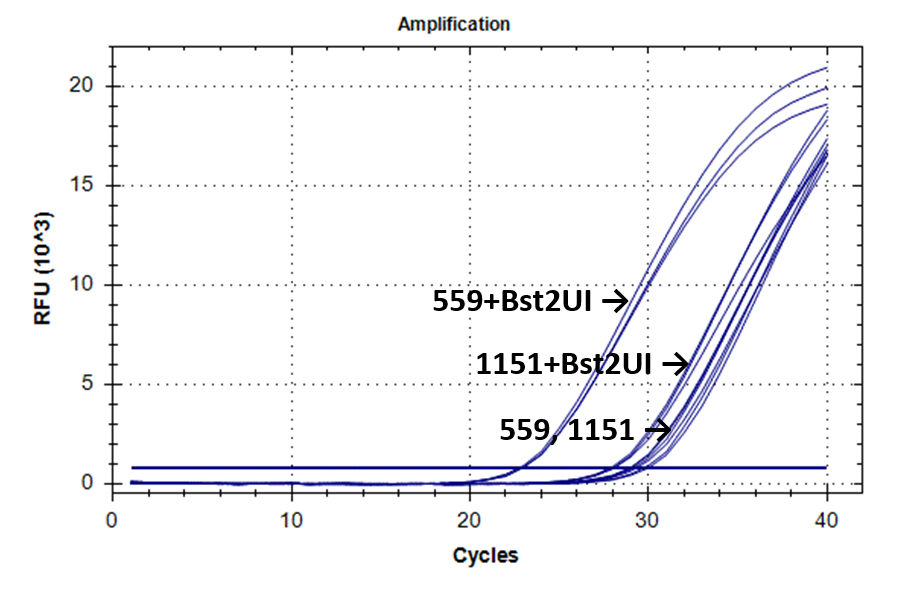 |
В таблице 2 представлены результаты ПЦР РВ 4-х фрагментов генома человека в 16-ти препаратах ДНК крови доноров.
Таблица 2
Значения Cq, полученные в ПЦР РВ 4-х выбранных фрагментов генома человека в 16-ти препаратах ДНК крови доноров. Синим фоном выделены номера препаратов ДНК, полученные фенольной очисткой.
|
№ |
9-604 + HinfI |
9-604 |
17-027 + MspI
|
17-027 |
11-215 + MspI |
11-215 |
1-178 + Bst2UI |
1-178 |
|
232 |
22,27±0,05 |
22,30±0,09 |
22,24±0,09 |
24,35±0,23 |
22,26±0,10 |
30,60±0,53 |
22,34±0,02 |
28,61±0,04 |
|
235 |
23,40±0,07 |
23,47±0,05 |
23,30±0,06 |
25,47±0,17 |
23,32±0,10 |
31,11±0,31 |
23,46±0,06 |
29,51±1,05 |
|
558 |
22,14±0,11 |
22,39±0,06 |
22,19±0,05 |
24,59±0,61 |
22,35±0,05 |
30,13±0,11 |
22,38±0,13 |
28,84±0,43 |
|
559 |
22,72±0,10 |
22,82±0,09 |
22,66±0,08 |
25,41±0,20 |
22,77±0,08 |
30,83±0,34 |
22,79±0,11 |
29,26±0,37 |
|
582 |
22,25±0,04 |
22,49±0,08 |
22,31±0,06 |
24,66±0,13 |
22,31±0,11 |
31,08±1,12 |
22,30±0,07 |
28,63±0,16 |
|
604 |
22,54±0,03 |
22,84±0,03 |
22,76±0,21 |
25,05±0,28 |
22,91±0,28 |
30,79±1,36 |
22,58±0,03 |
28,86±0,09 |
|
605 |
22,02±0,06 |
22,14±0,06 |
22,23±0,11 |
24,60±0,06 |
22,41±0,13 |
31,41±1,28 |
22,10±0,01 |
29,07±0,23 |
|
640 |
22,22±0,07 |
22,34±0,06 |
22,34±0,03 |
24,86±0,09 |
22,36±0,07 |
31,47±1,13 |
22,21±0,01 |
28,90±0,33 |
|
205 |
21,26±0,05 |
21,41±0,07 |
21,27±0,05 |
21,30±0,12 |
21,06±0,04 |
27,27±0,31 |
23,87±0,07 |
26,36±0,21 |
|
1086 |
21,87±0,05 |
21,96±0,06 |
22,02±0,17 |
22,11±0,06 |
21,99±0,09 |
28,35±0,38 |
27,94±0,27 |
28,99±0,17 |
|
1087 |
21,42±0,10 |
21,41±0,07 |
21,39±0,04 |
21,55±0,03 |
21,32±0,04 |
25,21±0,03 |
28,45±0,12 |
28,81±0,31 |
|
1137 |
21,46±0,06 |
21,60±0,18 |
21,56±0,06 |
21,70±0,10 |
21,65±0,10 |
27,21±0,11 |
27,36±0,37 |
28,99±0,17 |
|
1151 |
22,05±0,08 |
22,19±0,11 |
22,13±0,04 |
22,28±0,07 |
22,11±0,04 |
27,47±0,16 |
27,92±0,13 |
29,30±0,54 |
|
1205 |
21,41±0,09 |
21,54±0,08 |
21,54±0,09 |
21,65±0,08 |
21,74±0,28 |
27,13±0,13 |
26,71±0,29 |
28,00±0,16 |
|
1437 |
21,20±0,06 |
21,34±0,10 |
21,18±0,01 |
21,75±0,05 |
21,20±0,18 |
27,16±0,23 |
23,02±0,05 |
25,66±0,14 |
|
1487 |
21,84±0,06 |
22,09±0,06 |
22,00±0,02 |
22,39±0,06 |
21,34±0,15 |
27,66±0,28 |
24,66±0,22 |
27,04±0,35 |
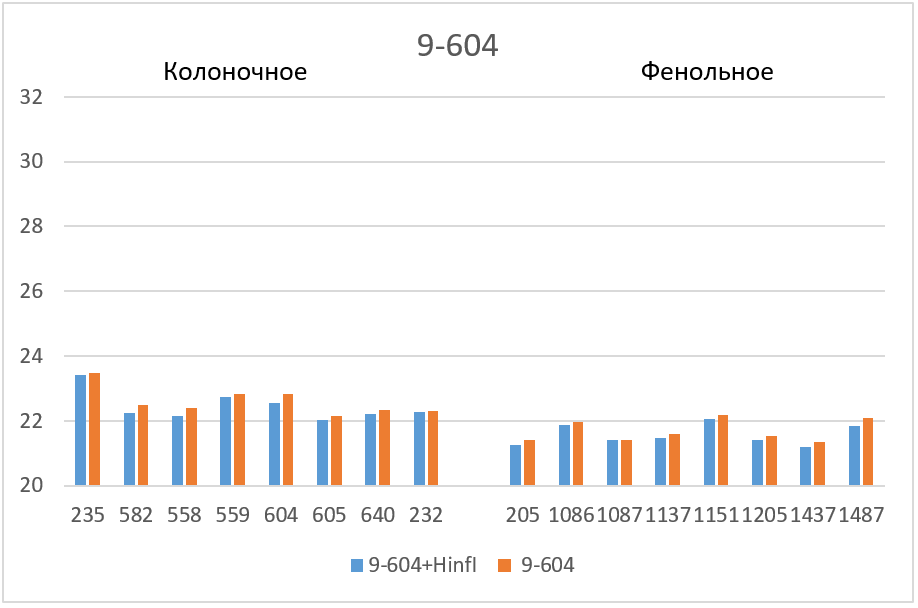
Как видно из таблицы 2 для каждого образца ДНК значения Cq, полученные при проведении ПЦР РВ на нативной ДНК либо больше, либо совпадают со значениями Cq, полученными при проведении ПЦР на гидролизованной ДНК. При этом, значения Cq для всех 16 образцов исходной ДНК в случае фрагмента 9-604 (столбик 3) и для 8-ми препаратов исходной ДНК после фенольной очистки в случае фрагмента 17-027 (нижняя половина столбика 5), а также значения Cq для образцов гидролизованной ДНК в случае всех 4-х фрагментов (за исключением фрагмента 1-178 на препаратах ДНК после фенольной очистки, столбики 2, 4, 6 и верхняя половина столбика 8) практически совпадают между собой для каждого из образцов ДНК. Эти близкие значения Cq для каждого образца ДНК (базовые значения), вероятно, соответствуют реальному значению Cq для искомого препарата ДНК, связанному обратно пропорциональной зависимостью с количеством анализируемых молекул геномной ДНК человека, в которой каждый из четырех изучаемых фрагментов представлен в одной копии.
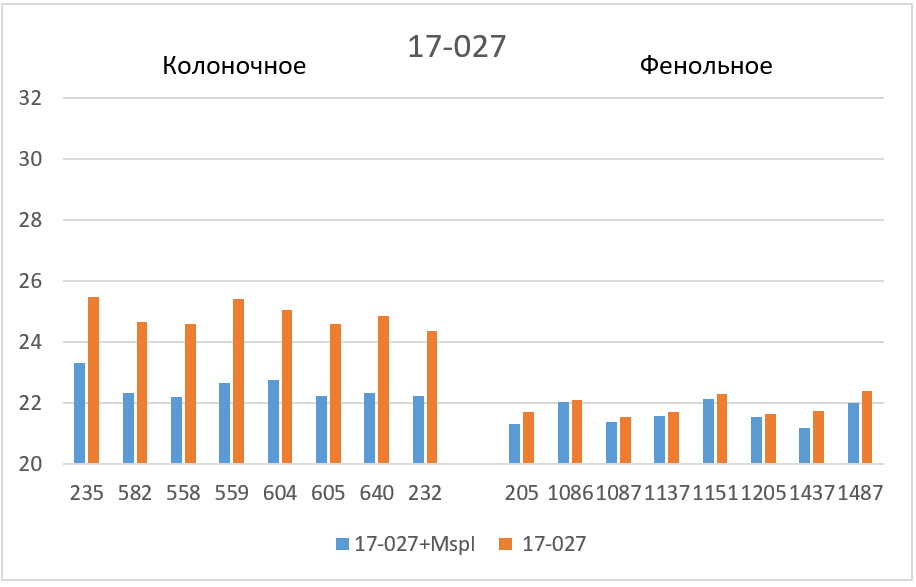
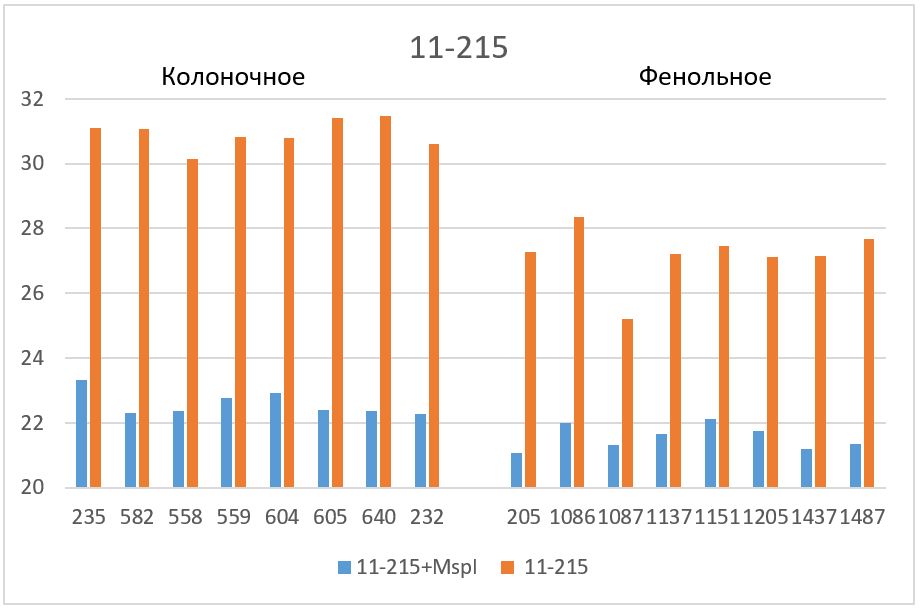
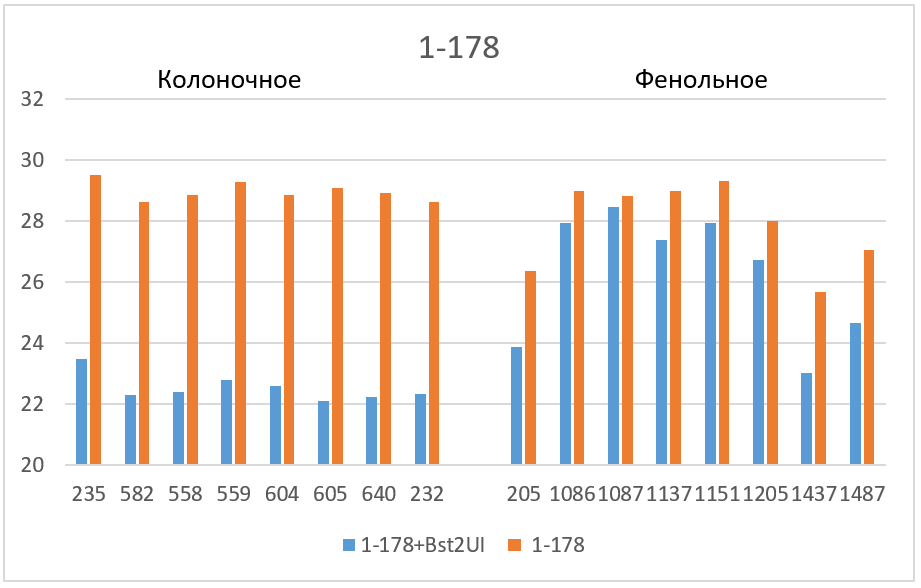
Завышенные значения Cq наблюдаются для трех фрагментов. 1) в случае фрагмента 17-027 значения Cq для препаратов нативной ДНК, очищенных на колонке, на 2 – 2,5 цикла больше, чем базовые значения (верхняя половина столбика 5); 2) в случае фрагмента 11-215 завышение Cq относительно базовых значений составляет для препаратов ДНК после фенольной очистки около 6 циклов, а для препаратов ДНК после колоночной очистки – около 8 циклов (столбик 7); и, наконец, 3) в случае фрагмента 1-178 наблюдается завышение Cq не только в случае всех препаратов нативной ДНК (столбик 9), но и в случае препаратов ДНК после фенольной очистки, которые гидролизовались рестриктазой Bst2UI (нижняя половина столбика 8).
На рисунке 3 полученные значения Cq представлены в виде диаграмм. Как видно из диаграмм после обработки ДНК рестриктазой, выщепляющей амплифицируемый фрагмент, значения Cq на всех фрагментах (кроме 1-178 в случае фенольной очистки) опускаются до базовых значений для каждого образца ДНК. Эффект завышения величины Cq при ПЦР на нативной ДНК может быть связан с влиянием соседних участков протяженных молекул ДНК, препятствующих амплификации, а также с возможными взаимодействиями протяженных молекул ДНК между собой. В дальнейшей работе мы планируем более детально изучить влияние участков ДНК вне ампликона на ПЦР РВ с TaqMan зондом.
Таким образом, при проведении ПЦР РВ необходимо использовать препараты ДНК, полученные методом очистки на колонке и предварительно обработанные рестриктазой, выщепляющей амплифицируемый фрагмент. ПЦР РВ препаратов ДНК, полученных фенольным методом, также как ПЦР РВ без обработки ДНК соответствующей рестриктазой, могут привести к получению завышенных значений Cq.
Литература
- А.Г. Акишев, Н.А. Нетесова, М.А. Абдурашитов, С.Х. Дегтярев Определение полиморфизма 5mC/T в повторе AluSx (CHR16: 75033884) в препаратах ДНК из крови человека методами GlaI- и FatI-ПЦР анализа // Эпигенетическая ДНК диагностика, DOI: 10.26213/SE.2021.11.76.001 (2021).
- А.Г. Акишев, Н.А. Нетесова, М.А. Абдурашитов, С.Х. Дегтярев Определение полиморфизма 5mC/T в позиции Chr1: 245618129 в препаратах ДНК из крови человека методами GlaI- и FatI-ПЦР анализа // Эпигенетическая ДНК диагностика, DOI: 10.26213/SE.2019.69.42836 (2021).
- А.Г. Акишев, Н.А. Нетесова, М.А. Абдурашитов, И.В. Вихлянов,М.К. Никитин, А.Б. Карпов, С.Х. Дегтярев Оценка риска возникновения рака молочной железы и рака желудка путем определения вариантов полиморфизма 5mC/T в интроне гена KIF26B в положении хр1: 245618129 // Эпигенетическая ДНК диагностика, DOI: 10.26213/SE.2019.78.40307 (2021).
- EV Dubinin, AG Akishev, MA Abdurashitov, SB Oleynikova, VL Sitko, and S Kh Degtyarev Real time GlaI-PCR assay of regulation regions of human genes HDAC4, RARB and URB1 // Research Journal of Pharmaceutical, Biological and Chemical Sciences, vol 7(2), pp. 667-676 (2016).
- Инструкция к набору для очистки ДНК из легкой фракции клеток крови // https://sibenzyme.com/wp-content/uploads/k008s-instruction-manual_rus_v.1.2-1.pdf
- Системы ПЦР реального времени с флуоресцентной детекцией CFX96. Руководство по эксплуатации // https://www.bio-rad.com/webroot/web/pdf/lsr/russian_federation/russian/literature/10021337_ru.pdf
- Набор для очистки ДНК из легкой фракции клеток крови // https://sibenzyme.com/product/k008/