Новая сайт-специфическая 5mC-зависимая ДНК-эндонуклеаза MoxI эффективно гидролизует ДНК последовательность 5’-R(5mC)GY-3’
В.А. Чернухин, В.С. Дедков, Д.А. Гончар, В.Н. Баймак, О.А. Беличенко, С.Х. Дегтярев
В.А. Чернухин, В.С. Дедков, Д.А. Гончар, В.Н. Баймак, О.А. Беличенко, С.Х. Дегтярев.
ООО «СибЭнзайм», Новосибирск
* автор для переписки: С.Х. Дегтярев, ООО «СибЭнзайм», ул. Тимакова, д. 2/12, Новосибирск, 630117, Россия; E-mail: degt@sibenzyme.com
РЕЗЮМЕ
Обнаружен штамм бактерии Microbacterium oxydans являющийся продуцентом 5-метилцитозин-зависимой сайт-специфической ДНК-эндонуклеазы MoxI, которая узнает и расщепляет в ДНК последовательность 5’-R(5mC)↓GY-3’/3’YG↓(5mC)R-5’. Ген moxI клонирован в составе вектора E.coli, получен препарат рекомбинантного фермента и изучены его свойства. Показано, что сайт узнавания ДНК-эндонуклеазы MoxI совпадает с сайтом узнавания 5mC-зависимой сайт-специфической ДНК-эндонуклеазы GlaI. Однако новый фермент, в отличие от GlaI, проявляет максимальную активность при 37°С и с большей эффективностью расщепляет последовательность узнавания с двумя 5-метилцитозинами.
Список сокращений: 5mC – 5-метилцитозин, R – пурин, Y – пиримидин, MD-эндонуклеаза – метилзависимая сайт-специфическая ДНК-эндонуклеаза, BSA – бычий сывороточный альбумин, IPTG – Изопропил-бета-D-тиогалактопиранозид, ДМСО – диметилсульфоксид, ДТТ – дитиотрейтол, ПААГ – полиакриламидный гель, п.н. – пары нуклеотидов, Трис – трис-(оксиметил)-аминометан, ЭДТА – этилендиаминтетрауксусная кислота.
Ключевые слова: штамм-продуцент, метилзависимая ДНК эндонуклеаза, эпигенетика
DOI: 10.26213/3034-4301.2025.6.3.001
Данные для цитирования:
В.А. Чернухин, В.С. Дедков, Д.А. Гончар, В.Н. Баймак, О.А. Беличенко, С.Х. Дегтярев (2025) Новая сайт-специфическая 5mC-зависимая ДНК-эндонуклеаза MoxI эффективно гидролизует в ДНК последовательность 5’-R(5mC)GY-3’ // ДНК-узнающие ферменты, том 2025(3), DOI: 10.26213/3034-4301.2025.6.3.001

Эта статья доступна по лицензии Creative Commons Attribution-NonCommercial-NoDerivatives 4.0 International
ВВЕДЕНИЕ
Метилзависимыми сайт-специфическими ДНК-эндонуклеазами, или MD-эндонуклеазами (MD – methyl dependent (англ.)) являются ферменты, которые узнают и расщепляют определенные метилированные последовательности ДНК и не гидролизуют немодифицированную ДНК [1]. По своим свойствам эти ферменты похожи на хорошо изученные эндонуклеазы рестрикции, поскольку подобно им они не требуют кофакторов, помимо ионов Mg2+, для проявления активности. Ранее открытый нами фермент GlaI, выделенный из бактериального штамма Glacial ice bacterium, является MD-эндонуклеазой, которая узнает в природной ДНК последовательность 5’-R(5mC)GY-3’/3’YG(5mC)R-5’ и гидролизует обе цепи ДНК посередине сайта с образованием «тупых» концов [2]. ДНК-метилтрансфераза DNMT3 млекопитающих и человека метилирует ДНК de novo с образованием последовательности 5’-R(5mC)GY-3’/3’YG(5mC)R-5’ [3], которая является сайтом узнавания GlaI. Вследствие этого фермент GlaI нашел применение в эпигенетической ДНК-диагностике и используется для определения статуса метилирования отдельных сайтов или фрагментов ДНК [4,5], а также для установления эпигенома человека [6]. В данной работе описано получение новой MD-эндонуклеазы MoxI и установлены оптимальные условия гидролиза ДНК данным ферментом, а также и проведено сравнение свойств MoxI и GlaI.
МАТЕРИАЛЫ И МЕТОДЫ
В работе использовались реактивы производства “Sigma-Aldrich” (США), “Thermo Fisher” (США), “Panreac” (Испания), “Диа-М” и “Хеликон” (Россия). Для хроматографической очистки фермента применялись следующие носители: фосфоцеллюлоза Р-11 («Whatman», Англия), фенил-сефароза и гепарин-сефароза («Sigma-Aldrich», США). Для выращивания бактериальных клеток использовались компоненты питательной среды фирмы ”Organotechnie” (Франция). Для экспериментов брали эндонуклеазы рестрикции, полинуклеотидкиназу фага Т4, реакционные SE-буферы «B100», «B», «G», «O», «W», «Y», «ROSE» производства ООО “СибЭнзайм” (Россия). В качестве маркёров молекулярных весов ДНК при получении электрофореграмм в агарозном и полиакриламидном гелях использовали ДНК-маркёры 1 kb, 100 bp, pUC19/MspI («СибЭнзайм», Россия). Для разбавления препаратов рекомбинатных GlaI и MoxI при измерении их активности использовался SE-буфер для хранения и разбавления ферментов «B100» (10 мМ Трис-HCl (pH 7,6), 50 мМ KCl, 0,1 мМ ЭДТА, 200 мкг/мл BSA, 1 мМ ДТТ, 50% глицерин). В работе использовали плазмидные ДНК pMTL22 [7], pUC19 [8] и pHspAI2 [9].
Эксперименты по анализу активности и субстратной специфичности рекомбинантного MoxI проводили в 20 или 50 мкл реакционной смеси, содержащей SE-буфер Y (33 мМ Трис-ацетат (pH 7,9), 10 мМ магния ацетат, 66 мМ калия ацетат, 1 мМ ДТТ) и 1 мкг субстратной ДНК при температуре 37°С в течение 1 часа, после чего проводили электрофорез в ПААГ. При тестировании активности MoxI в хроматографическом профиле аликвоты по 1 мкл из фракций добавляли к 20 мкл реакционной смеси, содержащей 1 мкг ДНК pHspAI2/GsaI в SE-буфере Y, инкубировали смесь в течение 15 мин при 37oС и проводили электрофорез в агарозном геле. При тестировании активности очищенного препарата MoxI брали 1 мкл неразбавленного фермента или 0,5, 1 и 2 мкл препарата MoxI, разбавленного в 10 раз SE-буфером «B100». Фермент добавляли к 50 мкл реакционной смеси, содержащей 1 мкг ДНК pHspAI2/GsaI и один из шести реакционных буферов для эндонуклеаз рестрикции (B, G, O, W, Y, ROSE). Смесь инкубировали в течение 1 ч при 25, 30, 37 или 55oС, после чего реакцию останавливали стоп-буфером (40% сахароза, 0,1 М ЭДТА, 0,05% бромфеноловый синий) и проводили электрофорез в агарозном геле.
Морфологические и физико-биохимические свойства исходной бактерии-продуцента определяли с использованием методик [10]. Определение рода микроорганизма проводили и по результатам секвенирования ПЦР-фрагмента гена 16S рРНК и по определителю Берджи [11].
Для определения места гидролиза межнуклеотидных связей ферментом MoxI использовали два [32P]-меченых ДНК-дуплекса (G3/G4 и G3/G2), содержащих сайт узнавания целевого фермента и сайты узнавания контрольных рестриктаз. Одну из цепей дуплексов (G3) модифицировали по 5’-концу с помощью Т4-полинуклеотидкиназы и γ[32P]ATP. После очистки олигонуклеотида от побочных продуктов реакции к нему добавляли комплементарный немеченый олигонуклеотид G4 (для GlaI и MoxI) или G2 (для рестриктаз HspAI и AspLEI). Пробирки прогревали 5 минут при 95°С с последующим охлаждением до комнатной температуры на рабочем столе. Реакцию гидролиза проводили в 10 мкл реакционной смеси, содержащей SE-буфер Y (+20% ДМСО в случае GlaI) или O, олигонуклеотидный дуплекс в концентрации 62.5 нМ и 1 мкл одного из ферментов. Инкубировали смесь течение 1 часа. Гидролиз GlaI проводили при температуре 25°С, а MoxI при температуре 37°С в SE-буфере Y. В контрольную реакционную смесь к дуплексу добавляли SE-буфер Y и 1 мкл рестриктазы HspAI, либо SE-буфер O и 1 мкл рестриктазы AspLEI, инкубацию проводили при 37°С. Продукты гидролиза наносили на 20% ПААГ с 8 М мочевиной и проводили электрофорез в Трис-боратном буфере. Для разделения продуктов ферментативного гидролиза меченых олигонуклеотидов применяли электрофорез в 20% ПААГ, содержавшем 8 М мочевину.
Рекомбинантный штамм E.coli ER2267(pMTL-MoxI), несущий плазмиду pMTL22 с клонированным геном moxI, нами был получен с использованием стандартных генно-инженерных методик [12]. Штамм Esherichia coli ER2267, использованный для клонирования гена изучаемого фермента, получен из “New England Biolabs” (США).
Наработка биомассы рекомбинантного штамма E.coli ER2267(pMTL22-MoxI), несущего плазмиду с геном MoxI
Для получения биомассы одиночную колонию штамма E.coli ER2267(pMTL22-MoxI) засевали во флакон со 100 мл питательной среды LB (1% триптон, 0,5% дрожжевой экстракт, 0,5% NaCl, рН 7,5) с ампициллином (50 мкг/мл). Культуру подращивали в течение ночи в термостате при 37°С. Затем полученный инокулят засевали по 5 мл в 12 флаконов со 100 мл среды LB с ампициллином. Культуру подращивали в течение 5 часов в термостатированной качалке при 30°С с перемешиванием при 140 об/мин. Затем в культуру добавляли IPTG до концентрации 1 мМ и растили еще в течение 20 часов. Затем 1 мл культуральной жидкости отбирали для анализа, а остальную культуру откручивали на центрифуге J2-21 («Beckman», США) при 6000 об/мин в течение 30 мин. Полученную биомассу клеток замораживали при -20°С.
Выделение рекомбинантной MD—эндонуклеазы MoxI
Условия проведения выделения и используемые буферы. Выделение проводили при +4оС с использованием растворов:
- буфер А — 10 мМ Трис-HCl, pH 7,5; 0,1 мМ ЭДТА и 7 мМ β-меркаптоэтанол;
- буфер Б – 60 мМ Трис-HCl, pH 7,5; 0,1 мМ ЭДТА и 7 мМ β-меркаптоэтанол.
Экстрагирование. 2,3 г биомассы E.coli ER2267(pMTL-MoxI) суспендировали в 25 мл буфера А, содержащего 0,1 M КCl, 1 мг/мл лизоцима, 0,1 мМ PMSF (фенилметилсульфонилфторид – ингибитор протеаз). После 60 мин перемешивания на магнитной мешалке суспензию в стеклянном стакане на 50 мл термостатировали в ледяной бане и затем клетки разрушали ультразвуком при максимальной мощности с амплитудой 22–24 мкм на приборе Soniprep 150 («MSE», Англия) с насадкой диаметром 2 см шестью импульсами по 30 сек с интервалом 2 мин. Экстракт осветляли на центрифуге J2-М1 («Beckman», США) в течение 40 мин в роторе JA-20 при 15000 об/мин.
Хроматография на фосфоцеллюлозе Р-11. Осветленный экстракт наносили на колонку с фосфоцеллюлозой P-11 объёмом 50 мл, предварительно уравновешенной буфером А с 0,05 М КCl. Колонку промывали 100 мл буфера А с 0,05 М КCl. Сорбированный материал элюировали линейным градиентом концентрации КCl 0,1–0,4 М в буфере А объемом 700 мл. Собирали 70 фракций по 10 мл. Объединяли фракции (29-35), содержащие максимум активности MoxI.
Хроматография на фенил-сефарозе. К 70 мл активной фракции добавляли 1 М Трис-HCl, pH 7,5 до концентрации 60 мМ (4,2 мл), сульфат аммония до концентрации 1,7 М (15,7 г) и наносили на колонку с 5 мл фенил-сефарозы, уравновешенной буфером Б с 1,7 М сульфата аммония. Колонку промывали 10 мл буфера Б c 1,7 M сульфата аммония. Фермент элюировали линейным градиентом 1,7–0 М сульфата аммония объемом 600 мл. Собирали 60 фракций по 10 мл. Объединяли фракции (37-43) с максимальной активностью фермента.
Хроматография на гепарин-сефарозе. 70 мл полученной фракции диализовали против 3 л буфера А в течение 16 часов и наносили на колонку с 5 мл гепарин-сефарозы, уравновешенной буфером А. После нанесения колонку промывали 10 мл буфера А. Фермент элюировали линейным градиентом концентрации КCl 0–0,15 М в буфере А, объемом 400 мл. Собирали 50 фракций по 8 мл. Фракции (18-28), содержащие максимальную активность MoxI, объединяли.
Концентрирование и хранение препарата
Полученные фракции с активностью MoxI диализовали против 900 мл концентрирующего буфера (10 мМ Трис-HCl (pH 7,5), 0,1 мМ ЭДТА, 7 мМ β-меркаптоэтанол, 0,2 М КCl, 50% глицерин) с ВSA в концентрации 200 мкг/мл, при перемешивании на магнитной мешалке в течение 16 ч. Препарат хранили при -20°C.
РЕЗУЛЬТАТЫ И ОБСУЖДЕНИЕ
Характеристика штамма-продуцента
Исследуемый бактериальный штамм был нами выделен из пробы пресной воды в ходе поиска микроорганизмов – продуцентов эндонуклеаз рестрикции и MD-эндонуклеаз. На основании морфолого-биохимических данных и анализа последовательности 16S рРНК (данные не приводятся) штамм идентифицировали как Microbacterium oxydans. Обнаруженный фермент был назван MoxI согласно общепринятой номенклатуре. Рекомбинантный штамм E.coli ER2267(pMTL-MoxI), несущий плазмиду с геном moxI, нами был получен путем клонирования ПЦР-фрагмента в вектор pMTL22.
Получение препарата фермента MoxI и изучение его свойств
В результате наработки культуры клеток рекомбинантного штамма E.coli ER2267(pMTL22-MoxI) выход биомассы составил 2,0±0,5 г/л. После хроматографической очистки из 2,3 г биомассы было получено 15 мл препарата MD-эндонуклеазы MoxI с концентрацией 5000 ед/мл. За одну единицу активности принимали минимальное количество фермента, способное полностью гидролизовать 1 мкг ДНК плазмиды pHspAI2/GsaI по сайту 5’-GCGC-3’ в 50 мкл реакционной смеси в оптимальном буфере при 37oС за 1 час.
Анализ субстратной специфичности рекомбинантного MoxI
Активность и субстратную специфичность препарата MD-эндонуклеазы MoxI определяли, взяв в качестве субстрата ДНК плазмиды с C5-метилированными цитозинами – pHspAI2 [9], которая содержит метилированные сайты 5’-G(5mC)GC-3’ и один сайт 5’-G(5mC)G(5mC)-3’, линеаризованной рестриктазой GsaI (pHspAI2/GsaI).
На рисунке 1А приведена электрофореграмма разделения продуктов гидролиза ДНК плазмиды pHspAI2/GsaI в SE-буфере Y при 37oС ферментами GlaI и MoxI, по-отдельности и совместно. На рисунке 1Б в качестве контроля специфичности приведены результаты тестирования препарата MoxI на ДНК фагов Лямбда и Т7, в которых отсутствуют метилированные сайты 5’-G(5mC)GC-3’.
Рисунок 1a
Электрофореграмма разделения продуктов гидролиза ДНК pHspAI2/GsaI препаратами ферментов GlaI и MoxI и расчетная картина распределения фрагментов расщепления этой ДНК по сайтам 5’-GCGC-3’
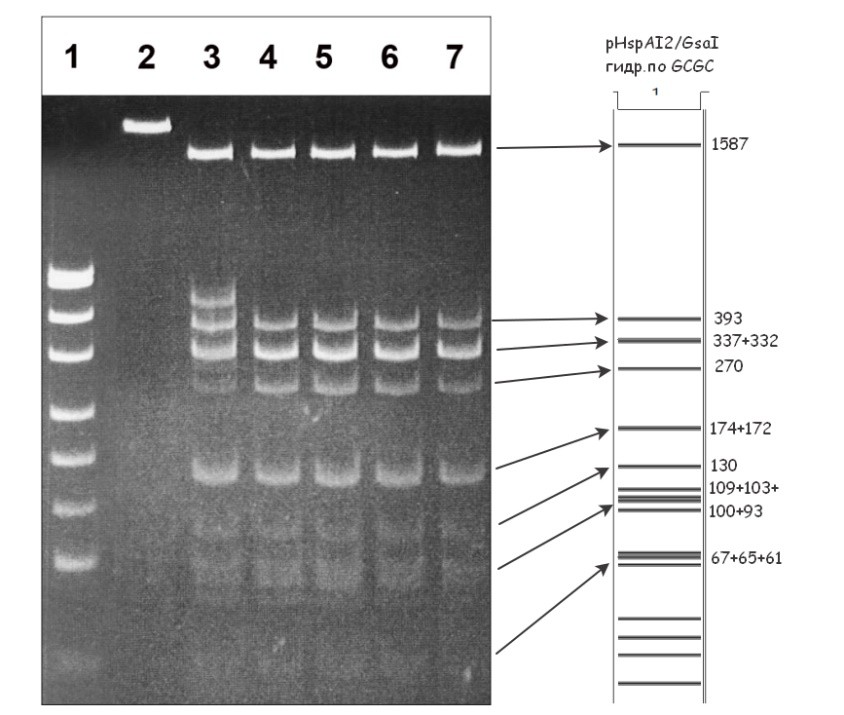
Дорожки:
- ДНК-маркер pUC19/MspI (от 26 до 501 п.н.);
- Исходная ДНК pHspAI2/GsaI;
- ДНК pHspAI2/GsaI + 1 мкл препарата GlaI;
- ДНК pHspAI2/GsaI + 1 мкл препарата GlaI + 20% ДМСО;
- ДНК pHspAI2/GsaI + 1 мкл препарата MoxI;
- ДНК pHspAI2/GsaI + 1 мкл препарата GlaI и 1 мкл препарата MoxI;
- ДНК pHspAI2/GsaI + 1 мкл препарата GlaI и 1 мкл препарата MoxI + 20% ДМСО.
Продукты расщепления разделены в 5 % ПААГ в TAE-буфере. Справа приведена теоретически рассчитанная картина гидролиза плазмиды pHspAI2/GsaI по сайтам 5’-GCGC-3’ с указанием длин получаемых фрагментов (п.н.).
Рисунок 1б
Электрофореграмма, полученная после обработки ДНК фагов Лямбда и Т7 препаратом фермента MoxI в SE-буфере Y при 37oС.
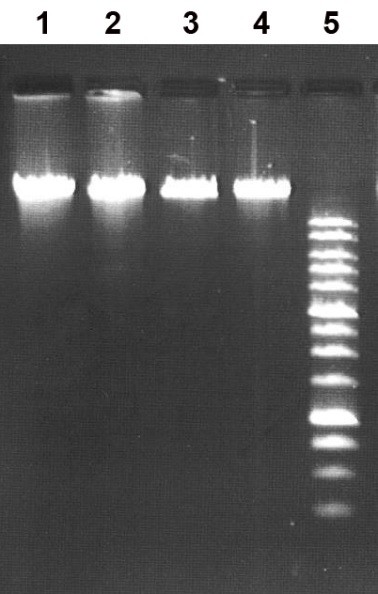
Дорожки:
- Исходная ДНК фага Лямбда;
- ДНК фага Лямбда + 1 мкл препарата MoxI;
- Исходная ДНК фага Т7;
- ДНК фага Т7 + 1 мкл препарата MoxI;
- 1 kb ДНК-маркеры молекулярного веса (от 0,25 до 10 т.п.н.).
Продукты расщепления разделены в 1% агарозном геле в TAE-буфере.
Как видно на рисунке 1А, при гидролизе ДНК pHspAI2/GsaI MD-эндонуклеазой GlaI с 20% ДМСО, гидролизе MoxI и совместном гидролизе двумя ферментами (дор.4, 5, 6 и 7) никаких изменений в картине распределения фрагментов не возникает. Отсюда можно сделать однозначный вывод о том, что оба фермента расщепляют данную метилированную плазмиду по сайту 5’-G(5mC)GC-3’. При этом, в случае GlaI степень гидролиза субстрата без добавления ДМСО (дор. 3) меньше по сравнению с гидролизом MoxI (дор. 5), т.е. новый фермент эффективнее гидролизует узнаваемую последовательность в случае метилирования только двух внутренних 5-метилцитозинов. А для GlaI, как было показано ранее [13], для получения полного гидролиза субстрата с двумя 5-метилцитозинами требуется добавление 20% ДМСО или присутствие в сайте 3-х либо 4-х 5-метилцитозинов. Активность же MoxI не зависит от присутствия ДМСО (дор. 5 и 7), и он одинаково эффективно расщепляет ДНК с двумя, тремя и четырьмя 5-метилцитозинами.
Таким образом, представленное на рисунке 1А сравнение полученной с помощью MoxI картины гидролиза ДНК плазмиды pHspAI2/GsaI с теоретически рассчитанными данными расщепления ее последовательности по сайтам сайт-специфических эндонуклеаз позволяет сделать вывод, что фермент MoxI гидролизует данный субстрат по сайту 5’-G(5mC)GC-3’, и является, таким образом, изошизомером MD-эндонуклеазы GlaI, изученной нами ранее [2, 13]. При этом, новый фермент, так же как и GlaI, не расщепляет ДНК, в которой отсутствуют метилированные последовательности 5’-G(5mC)GC-3’ (Рисунок 1Б, дорожки 2 и 4).
Далее мы проверили способность MD-эндонуклеазы MoxI расщеплять не только сайт 5’-G(5mC)GC-3’, но и все последовательности ДНК 5’-R(5mC)GY-3’, которые узнаются ферментом GlaI. Для этого мы использовали плазмиду pUC19, линеаризованную рестриктазой PstI и обработанную ДНК-метилтрансферазой SssI, образующей метилированные последовательности 5’-(5mC)G-3’. Данный субстрат обрабатывали ферментами GlaI и MoxI, по-отдельности и совместно. На рисунке 2 представлена электрофореграмма разделения продуктов гидролиза после проведения электрофореза в 7% ПААГ.
Рисунок 2
Электрофореграмма разделения продуктов гидролиза ДНК pUC19/PstI+M.SssI препаратами ферментов GlaI и MoxI и расчетная картина распределения фрагментов расщепления этой ДНК по сайтам 5’- RCGY -3’
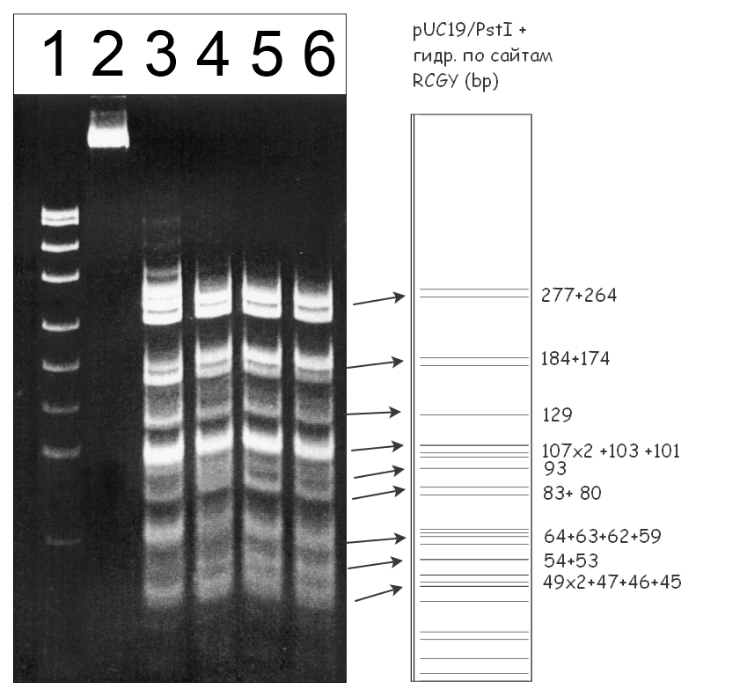
Дорожки:
- ДНК-маркер pUC19/MspI (от 26 до 501 п.н.);
- Исходная ДНК pUC19/PstI+M.SssI;
- ДНК pUC19/PstI+M.SssI + 1 мкл препарата GlaI;
- ДНК pUC19/PstI+M.SssI + 1 мкл препарата GlaI + 20% ДМСО;
- ДНК pUC19/PstI+M.SssI + 1 мкл препарата MoxI;
- ДНК pUC19/PstI+M.SssI + 1 мкл препарата GlaI и 1 мкл препарата MoxI;
Продукты расщепления разделены в 7 % ПААГ. Справа приведена теоретически рассчитанная картина гидролиза плазмиды pUC19/PstI по сайтам 5’-RCGY-3’.
Из рисунка видно, что, как и в случае гидролиза плазмиды pHspAI2/GsaI, картины расщепления ДНК pUC19/PstI+M.SssI MD-эндонуклеазами GlaI+ДМСО и MoxI одинаковы (дор. 4 и 5), а при совместном гидролизе субстрата двумя ферментами картина распределения фрагментов никак не меняется (дор. 6). Следовательно, можно сделать вывод, что новый фермент MoxI также узнает в ДНК последовательность 5’-R(5mC)GY-3’.
В ходе дальнейшей работы нами были определены оптимальные условия для работы MD-эндонуклеазы MoxI. Для этого использовали реакционную смесь, содержащую 1 мкг ДНК pHspAI2/GsaI, один из шести буферов для эндонуклеаз рестрикции (B, G, O, W, Y, ROSE), с добавлением 0,5, 1 или 2 мкл разбавленного в 10 раз SE-буфером «B100» препарата фермента MoxI, и четыре варианта температуры реакции – 25, 30, 37 и 55oС. Результаты приведены на рисунках 3, 4А и 4Б.
Рисунок 3
Электрофореграмма разделения продуктов гидролиза ДНК pHspAI2/GsaI препаратом MD-эндонуклеазы MoxI в шести SE-буферах при 37oС
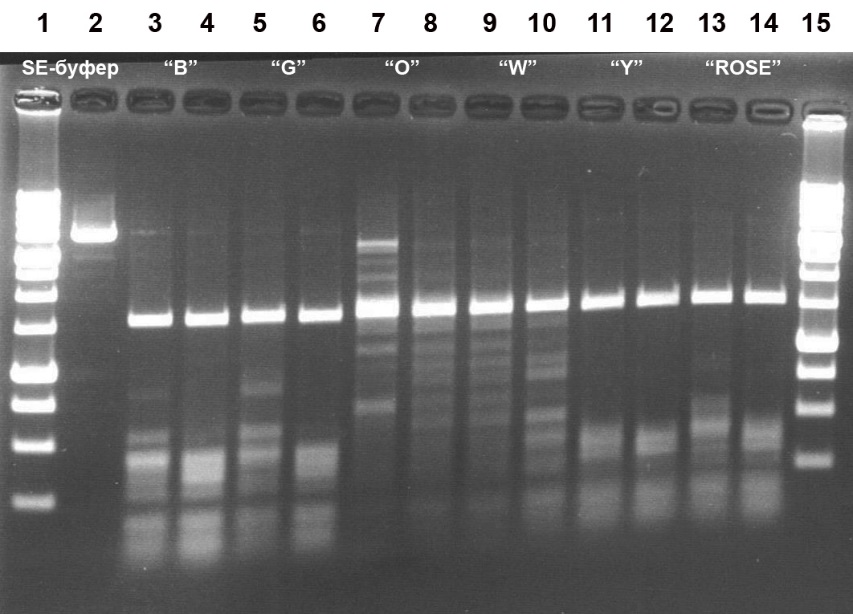
Дорожки:
2 – исходная ДНК pHspAI2/GsaI;
3, 5, 7, 9, 11, 13 – ДНК pHspAI2/GsaI + 1 мкл разбавленного в 10 раз препарата MoxI;
4, 6, 8, 10, 12, 14 – ДНК pHspAI2/GsaI + 2 мкл разбавленного в 10 раз препарата MoxI;
3 и 4 – SE-буфер B;
5 и 6 – SE-буфер G;
7 и 8 – SE-буфер O;
9 и 10 – SE-буфер W;
11 и 12 – SE-буфер Y;
13 и 14 – SE-буфер ROSE;
1 и 15 – 1 kb ДНК-маркеры молекулярного веса (от 0,25 до 10 т.п.н.).
Продукты расщепления разделены в 1,2% агарозном геле в TAE-буфере.
Рисунок 4а
Электрофореграмма разделения продуктов гидролиза ДНК pHspAI2/GsaI препаратом MD-эндонуклеазы MoxI при температурах 30, 37 и 55oС в SE-буфере Y.
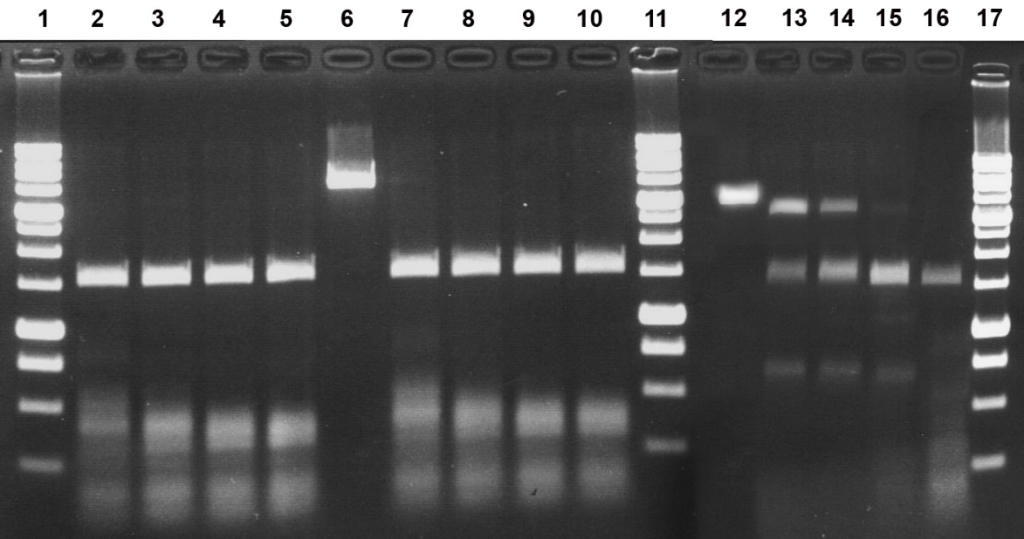
Дорожки:
2-5 – инкубация при 30oС;
7-10 – инкубация при 37oС;
13-16 – инкубация при 55oС;
6 и 12 – исходная ДНК pHspAI2/GsaI;
2, 7, 13 – ДНК pHspAI2/GsaI + 0,5 мкл разбавленного в 10 раз препарата MoxI;
3, 8, 14 – ДНК pHspAI2/GsaI + 1 мкл разбавленного в 10 раз препарата MoxI;
4, 9, 15 – ДНК pHspAI2/GsaI + 2 мкл разбавленного в 10 раз препарата MoxI;
5, 10, 16 – ДНК pHspAI2/GsaI + 1 мкл неразбавленного препарата MoxI;
1, 11, 17 – 1 kb ДНК-маркеры молекулярного веса (от 0,25 до 10 т.п.н.).
Продукты расщепления разделены в 1,2% агарозном геле.
Рисунок 4б
Электрофореграмма разделения продуктов гидролиза ДНК pHspAI2/GsaI препаратом MD-эндонуклеазы MoxI при температурах 25 и 30oС в SE-буфере Y
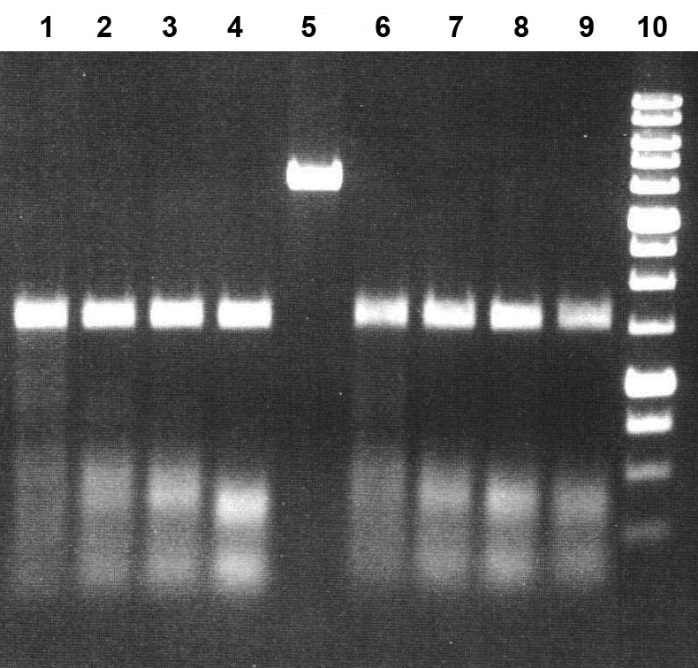
Дорожки:
1-4 – инкубация при 25oС;
6-9 – инкубация при 30oС;
5 – исходная ДНК pHspAI2/GsaI;
1 и 6 – ДНК pHspAI2/GsaI + 0,5 мкл разбавленного в 10 раз препарата MoxI;
2 и 7 – ДНК pHspAI2/GsaI + 1 мкл разбавленного в 10 раз препарата MoxI;
3 и 8 – ДНК pHspAI2/GsaI + 2 мкл разбавленного в 10 раз препарата MoxI;
4 и 9 – ДНК pHspAI2/GsaI + 1 мкл неразбавленного препарата MoxI;
10 – 1 kb ДНК-маркеры молекулярного веса (от 0,25 до 10 т.п.н.).
Продукты расщепления разделены в 1,2% агарозном геле.
Анализ полученных результатов позволил заключить, что MoxI проявляет максимальную активность в SE-буфере Y (33 мМ Трис-ацетат (pH 7,9), 10 мМ Mg-ацетат, 66 мМ K-ацетат, 1 мМ ДТТ) (рис. 3, дор. 11-12). Оптимальной температурой реакции является 37oС, хотя фермент не менее эффективно работает и при 30 oС (рис. 4А, дор. 2-5 и 7-10). При этих условиях активность фермента составляет не менее 5000 ед/мл. Активность MoxI при 55oС составляет не более 20% от оптимальной (рис. 4А, дор. 13-16). При 25oС активность фермента составляет не менее 50% от оптимальной (рис. 4Б, дор. 1-2). Необходимо отметить, что новый фермент оказался термостабильным, поскольку полной инактивации MoxI в реакционной смеси при нагревании до 65 или 80oС за 20 мин не происходит (данные не приведены).
В таблице 1 приведены данные по активности ферментов MoxI и GlaI в шести основных SE-буферах для эндонуклеаз рестрикции.
Таблица 1
Активность препаратов эндонуклеаз рестрикции MoxI и GlaI в шести основных SE-буферах для эндонуклеаз рестрикции (в % от максимальной).
| SE буфер | B | G | O | W | Y | ROSE |
| MoxI | 50–75 | 75–100 | 10–25 | 25–50 | 100 | 80 |
| GlaI | 75–100 | 75–100 | 25–50 | 25–50 | 100 | 100 |
Определение позиции гидролиза ДНК MD-эндонуклеазой MoxI
Чтобы определить место гидролиза ДНК новым ферментом мы использовали два двуцепочечных радиоактивно-меченых с 5’-конца олигонуклеотида (дуплекса) G3*/G4 и G3*/G2 (меченая цепь отмечена знаком *). Дуплекс G3*/G4 содержит два 5-метицитозина (по одному в каждой цепи) так, что образуется сайт узнавания GlaI (подчеркнут). Дуплекс G3*/G2 использовали в качестве контрольного, т.к. он отличается от первого только тем, что цепь G2 содержит неметилированную последовательность 5’-GCGC-3’, а сам дуплекс является полуметилированным и способен эффективно расщепляться рестриктазами HspAI [14] и AspLEI [15], узнающие и расщепляющие последовательность (5’-G(5mC)GC-3’/3’-CGCG-5’). Эти ферменты гидролизуют данный субстрат на расстоянии одного нуклеотида слева и справа, соответственно, от места гидролиза GlaI (см. Рис. 5А). Электрофореграмма, полученная после разделения продуктов расщепления ферментами дуплексов G3*/G4 и G3*/G2 в 20% ПААГ с 8 М мочевиной, представлена на Рис. 5Б.
Рисунок 5а
Структура олигонуклеотидных дуплексов G3*/G4 и G3*/G2 для определения позиции гидролиза ДНК MD-энлонуклеазой MoxI. Последовательность, узнаваемая ферментами, подчеркнута и выделена жирным. Предсказанные позиции гидролиза контрольными ферментами показаны косыми стрелками. Реальные точки расщепления показаны вертикальными стрелками.
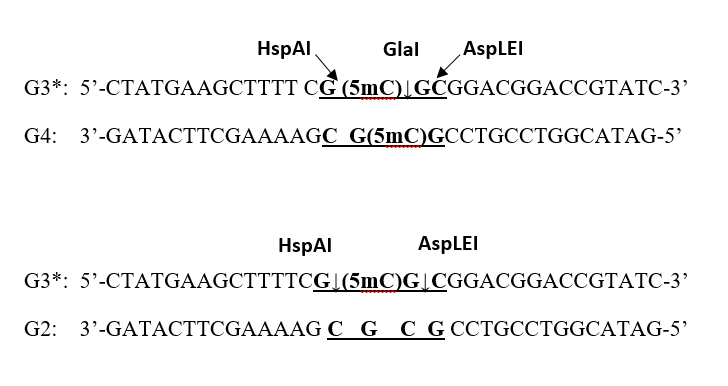
Рисунок 5б
Определение позиции гидролиза MoxI на олигонуклеотидном дуплексе G3*/G4. Электрофореграмма получена в 20% ПААГ с 8 М мочевиной после разделения продуктов расщепления ферментами GlaI или MoxI дуплекса G3*/G4, а также HspAI или AspLEI – дуплекса G3*/G2.
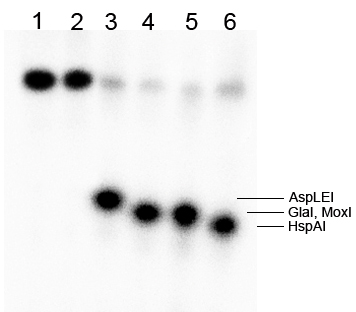
Дорожки:
- исходный дуплекс G3*/G4;
- исходный дуплекс G3*/G2;
- дуплекс G3*/G2 + 1 мкл AspLEI;
- дуплекс G3*/G4 + 20% ДМСО + 1 мкл GlaI;
- дуплекс G3*/G4 + 1 мкл MoxI;
- дуплекс G3*/G2 + 1 мкл HspAI.
Из рисунка видно, что позиция гидролиза MoxI совпадает с позицией гидролиза GlaI (отмечена стрелкой): 5’-G(5mC)↓GC-3’. Таким образом, MoxI образует в результате гидролиза ДНК «тупые» концы и является истинным изошизомером GlaI. Эта же авторадиограмма геля подтверждает результат, полученный ранее на плазмидных ДНК: MD-эндонуклеаза MoxI способна эффективно расщеплять ДНК-субстрат с двумя внутренними 5-метилцитозинами (дор. 5), тогда как в случае GlaI эффективный гидролиз такого субстрата происходит только в присутствии в реакционной смеси ДМСО (дор. 4).
Новая MD-эндонуклеаза MoxI, являясь изошизомером GlaI, используется в эпигенетических исследованиях, в частности при проведении GlaI- и GLAD-ПЦР анализа [14, 15], поскольку в отличие от ранее используемого для этих целей GlaI, одинаково хорошо работает при температурах 30 и 37oС и эффективно расщепляет сайт 5’-R(5mC)GY-3’ с двумя внутренними 5-метилцитозинами. Это позволяет добиться более полного гидролиза анализируемых образцов ДНК в отсутствие ДМСО, который необходим для полного расщепления ДНК ферментом GlaI, но при этом ингибирует ПЦР.
Список литературы
- Метилзависимые ДНК эндонуклеазы // https://sibenzyme.com/product-category/md/
- Tarasova G. V., Nayakshina T. N., Degtyarev S. Kh. 2008. Substrate specificity of new methyl-directed DNA endonuclease GlaI. BMC Molecular Biology, 9:7.
- Handa V., Jeltsch A. 2005. Profound flanking sequence preference of Dnmt3a and Dnmt3b mammalian DNA methyltransferases shape the human epigenome. J. Mol. Biol., 348 (5): 1103-1112.
- Гончар Д.А., Акишев А.Г., Дегтярев С.Х. 2010. BlsI- и GlaI- ПЦР анализ – новый метод исследования метилированных участков ДНК. Вестник биотехнологии и физико-химической биологии имени Ю. А. Овчинникова, Т.6, № 1, 5-12.
- Кузнецов В.В., Абдурашитов М.А., Акишев А.Г., Дегтярев С.Х. 2014. Способ определения нуклеотидной последовательности Pu(5mC)GPy в заданном положении протяженной ДНК. Патент на изобретение RU 2525710 С1.
- Abdurashitov M.A., Tomilov V.N., Gonchar D.A., Kuznetsov V.V., Degtyarev S.Kh. 2015. Mapping of R(5mC)GY Sites in the Genome of Human Malignant Cell Line Raji. Biol. Med. (Aligarh), V.7, Is.4. BM-135-15.
- Chambers S.P., Prior S.E., Barstow D.A., Minton N.P. 1998. The pMTLnic— cloning vectors. I. Improved pUC polylinker regions to facilitate the use of sonicated DNA for nucleotide sequencing. Gene, 68. 139-149.
- Yanisch-Perron C., Vieira J., Messing J. 1985. Improved M13 phage cloning vectors and host strains: nucleotide sequences of the M13mp18 and pUC19 vectors. Gene, 33. 103-119.
- Чернухин В.А., Гончар Д.А., Килева Е.В., Соколова В.А., Голикова Л.Н., Дедков В.С., Михненкова Н.А., Дегтярёв С.Х.2012. Новая метилзависимая сайт-специфическая ДНК-эндонуклеаза MteI расщепляет девятинуклеотидную последовательность 5`-G(5mC)G(5mC)NG(5mC)GC-3`/3`-CG(5mC)GN(5mC)G(5mC)G-5`. Вестник биотехнологии и физико-химической биологии имени Ю. А. Овчинникова, Т.8, №1. 16-26.
- Руководство к практическим занятиям по микробиологии. Под ред. Н.С. Егорова. М., 1995.
- Определитель бактерий Берджи. Под ред. Дж. Хоулта и др.: 9-е издание в 2-х томах. Пер. с англ. под ред. Г.А. Заварзина. М., 1997.
- Sambrook J., Fritsch E.F., Maniatis T. Molecular cloning: a laboratopy manual. 1987. Cold Spring Harbor Laboratory Press, USA.
- Abdurashitov M.A., Chernukhin V.A., Gonchar D.A., Dedkov V.S., Mikhnenkova N.A. and Degtyarev S.Kh. 2016. Dimethyl Sulfoxide Changes the Recognition Site Preference of Methyl- directed Site-specific DNA Endonuclease GlaI. Research Journal of Pharmaceutical, Biological and Chemical Sciences, 7(1). 1733-1739.
- Набор для GlaI-ПЦР анализа // https://sibenzyme.com/product/glai-pcr/
- Набор для GLAD-ПЦР анализа // https://sibenzyme.com/product/glad-hybrid/
