Эндонуклеаза рестрикции BmoI из штамма бактерии Brevundimonas mongoliensis 53 узнает последовательность ДНК 5’-GG^GWCCC-3’
Д.А. Гончар1, М.А. Абдурашитов1, В.А. Чернухин1, В.С. Дедков1, Н.А. Михненкова1, ВЕ. Рыжова1, С.Х. Дегтярёв1
1 ООО «СибЭнзайм», Новосибирск
* автор для переписки: Д.А. Гончар, ООО «СибЭнзайм», ул. Тимакова, д. 2/12, Новосибирск, 630117, Россия; тел.: +7(383)333-4991; факс: +7(383)333-6853; E-mail: gonchar@sibenzyme.ru
РЕЗЮМЕ
Нами выявлен бактериальный штамм Brevundimonas mongoliensis 53, который является продуцентом новой крупнощепящей эндонуклеазы рестрикции, названной BmoI. Фермент BmoI узнает семинуклеотидную палиндромную последовательность ДНК 5’-GG↓GWCCC-3’ (W = A или T) и расщепляет ее как указано стрелкой, образуя трехнуклеотидные
5’-выступающие «липкие» концы. BmoI является изошизомером ранее описанной эндонуклеазы рестрикции SanDI [1]. Штамм-продуцент идентифицировали по морфологическим и биохимическим признакам, а также по анализу первичной структуры фрагмента гена 16S рРНК. Препарат эндонуклеазы рестрикции BmoI с концентрацией 2000 е.а./мл был получен путем очистки в три хроматографические стадии. Оптимальными условиями работы фермента являются SE-буфер W (10 мМ Трис-HCl (pH 8,5), 10 мМ MgCl2, 100 мМ NaCl, 1 мМ ДТТ) и температура реакции 37°С.
Список сокращений: BSA – бычий сывороточный альбумин; ДТТ – дитиотреитол; Т7 ДНК – ДНК бактериофага T7; λ ДНК – ДНК бактериофага λ; е.а. – единицы активности; Трис – трис-(оксиметил)аминометан; ПААГ – полиакриламидный гель; п.н. – пар нуклеотидов; рестриктаза, ЭР – эндонуклеаза рестрикции; ЭДТА – этилендиамин-тетрауксусная кислота.
Ключевые слова: штамм-продуцент, выделение фермента, эндонуклеаза рестрикции, изошизомер
DOI: 10.26213/3034-4301.2025.5.2.002
Данные для цитирования:
Д.А. Гончар, М.А. Абдурашитов, В.А. Чернухин, В.С. Дедков, Н.А. Михненкова, В.Е. Рыжова, С.Х. Дегтярёв. (2025) Эндонуклеаза рестрикции BmoI из штамма бактерии Brevundimonas mongoliensis 53 узнает последовательность ДНК 5’-GG^GWCCC-3’. // ДНК-узнающие ферменты, том 2025(2), DOI: 10.26213/3034-4301.2025.5.2.002

Эта статья доступна по лицензии Creative Commons Attribution-NonCommercial-NoDerivatives 4.0 International
ВВЕДЕНИЕ.
Эндонуклеазы рестрикции II типа специфически расщепляют ДНК внутри или рядом с сайтом узнавания и широко применяются в молекулярной биологии, ДНК-диагностике и генно-инженерных работах. Данная работа посвящена описанию новой крупнощепящей эндонуклеазы рестрикции BmoI, узнающей семинуклеотидную палиндромную последовательность ДНК 5’-GG↓GWCCC-3’/3’-CCCWG↓GG-5’ и расщепляющей ее, как показано стрелками.
УСЛОВИЯ ЭКСПЕРИМЕНТА
В работе использовались реактивы производства “Sigma-Aldrich” (США), “Fisher” (Германия), “Panreac” (Испания), “Диа-М” и “Хеликон” (Россия). Для хроматографической очистки фермента применялись следующие носители: фосфоцеллюлоза P11, гепарин-сефароза (“Sigma”, США). Для выращивания бактериальных клеток использовались компоненты питательной среды фирмы ”Organotechnie” (Франция). Для экспериментов брали эндонуклеазы рестрикции, полинуклеотидкиназу фага Т4, препарат ДНК фага Т7, маркеры молекулярного веса ДНК, реакционные SE-буферы для эндонуклеаз рестрикции «B100», «B», «G», «O», «W», «Y», «ROSE» производства ООО “СибЭнзайм” (Россия).
Морфологические и физико-биохимические свойства штамма изучали с использованием методик [2]. Определение рода микроорганизма проводили по определителю Берджи [3] и по результатам секвенирования ПЦР-фрагмента гена 16S рРНК.
Тестирование рестриктазной активности BmoI.
В качестве субстрата для анализа активности BmoI использовали ДНК фага T7. При тестировании активности рестриктазы в хроматографическом профиле аликвоты по 1 мкл из фракций добавляли к 20 мкл реакционной смеси, содержащей 1 мкг ДНК фага T7 в SE-буфере W, инкубировали смесь в течение 15 мин при 37°С. Продукты реакции наносили на 1% агарозный гель и проводили электрофорез в Трис-ацетатном буфере (50 мМ Трис-ацетат (pH 8,0), 20 мМ Na-ацетат, 2 мМ ЭДТА) при напряжении 180V. После окрашивания бромистым этидием гель фотографировали в УФ-свете. За единицу активности рестриктазы принимали минимальное количество фермента, необходимое для полного расщепления 1 мкг ДНК фага T7 в SE-буфере W при 37°С в 50 мкл реакционной смеси за 1 час.
Чтобы определить место гидролиза межнуклеотидных связей в ДНК изучаемым ферментом, использовали олигонуклеотидный [32P]-меченый ДНК-дуплекс, содержащий сайт узнавания BmoI и сайты узнавания контрольных ферментов. Для разделения продуктов ферментативного гидролиза меченых олигонуклеотидов применяли электрофорез в 20% ПААГ, содержавшем 8 М мочевину.
Выращивание клеток штамма B. mongoliensis 53.
Штамм B. mongoliensis 53 выращивали в LM/4-бульоне, содержащем 0,25% триптон (Organotechnie, Франция), 0,13% дрожжевой экстракт (той же фирмы), 0,13% NaCl, 0,013% MgCl2 и 0,0003% тиамин. Рассевали на LM/4-агаре – 1,5% агар (C.E. Roeper GmbH, Германия) в LM/4-бульоне. Среды готовили по 0,3 л в 0,5-литровых конических колбах. Для получения биомассы культуру выращивали в колбах с бульоном при 28°С со встряхиванием 2 дня. Клетки собирали центрифугированием на центрифуге Beckman J2-MI (США) в роторе JA-10 при 4°С, 8000 об/мин в течение 20 мин. С 7 л получали 25 г биомассы, которую замораживали при -20°С.
Выделение эндонуклеазы рестрикции BmoI.
Условия проведения выделения и используемые буферы. Выделение проводили при +4°С с использованием растворов на основе буфера:
• буфер А — 10 мМ Трис-HCl pH 7,5; 0,1 мМ ЭДТА и 7 мМ β-меркаптоэтанол.
Экстрагирование. 10 г биомассы B. mongoliensis 53 суспендировали в 50 мл буфера А, содержащего 0,2 М NaCl, 1 мг/мл лизоцим, 0,1 мМ PMSF (фенилметил-сульфонилфторид – ингибитор протеаз). После 1 часа перемешивания на магнитной мешалке суспензию в стеклянном стакане на 100 мл термостатировали в ледяной бане и затем клетки разрушали ультразвуком при максимальной мощности с амплитудой 22–24 мкм на приборе Soniprep 150 («MSE», Англия) с насадкой диаметром 2 см десятью импульсами по 30 сек с интервалом 2 мин. Экстракт осветляли на центрифуге J2-М1 («Beckman», США) в течение 40 минут в роторе JA-20 при 15000 об/мин.
Хроматография на фосфоцеллюлозе Р-11. Экстракт наносили на колонку с 10 мл фосфоцеллюлозы P-11 и промывали 60 мл буфера А с 0,2 М NaCl. Сорбированный материал элюировали линейным градиентом концентрации NaCl от 0,2 до 0,6 М в буфере А, объёмом 600 мл. Собирали 60 фракций по 10 мл. Объединяли фракции, содержащие максимум целевой активности (№ 13–21), и диализовали против 1,5 л буфера А в течение 2 ч.
Хроматография на гепарин-сефарозе. Объединённую фракцию после диализа наносили на колонку с гепарин-сефарозой объёмом 4 мл, уравновешенной буфером А с 0,05 М NaCl. Колонку промывали 10 мл того же буфера. Сорбированный материал элюировали линейным градиентом концентрации NaCl от 0,05 до 0,5 М в буфере А, объёмом 120 мл, собирали 40 фракций по 3 мл. Фракции, содержащие максимум активности BmoI (№ 31–36), объединяли и диализовали против 0,8 л буфера А в течение 2 ч.
Рехроматография на фосфоцеллюлозе Р-11. Объединённую фракцию после диализа повторно наносили на колонку с фосфоцеллюлозой P-11 объёмом 10 мл, уравновешенной буфером А с 0,2 М NaCl. Колонку промывали 20 мл того же буфера. Сорбированный материал элюировали линейным градиентом NaCl от 0,2 до 0,6 М в буфере А, объёмом 120 мл. Собирали 40 фракций по 3 мл. Фракции, содержащие максимальную активность фермента (№ 15–21), объединяли.
Концентрирование и хранение препарата BmoI. Объединённую фракцию после диализа диализовали против 350 мл концентрирующего буфера (10 мМ Трис-HCl (pH 7,5), 0,05 М NaCl, 0,1 мМ ЭДТА, 7 мМ β-меркаптоэтанол, 50% глицерин) при перемешивании на магнитной мешалке в течение 16 ч. Препарат хранили при –20°С.
РЕЗУЛЬТАТЫ И ОБСУЖДЕНИЕ
Характеристика и таксономическая идентификация штамма.
Штамм выделен из образца почвы. На LM/4-агаре за 3 дня при 24°С образует круглые колонии до 1 мм в диаметре. Колонии выпуклые, блестящие, полупрозрачные, бледно-оранжевые. Клетки палочковидные, 0,3 × 2–3 мкм, неподвижные, грамотрицательные, слабокаталазоположительные, оксидазоположительные. Растут в аэробных условиях при температуре от 8 до 30°С и не растут при 37°С. Содержание G+C в ДНК штамма (67±5%) определяли по [4].
Для идентификации штамма по 16S рРНК из клеток штамма выделяли ДНК и проводили ПЦР гена 16S рРНК с использованием праймеров 5’-agagtttgatcmtggctca-3’ и 5’-tacggytaccttgttacgact-3’, представляющих модификации fDl и rPl [5]. Секвенировали 1425 нуклеотидов части гена 16S рРНК (Рис. 1).
Рисунок 1
Нуклеотидная последовательность фрагмента гена 16S рРНК (1425 п.н.) штамма-продуцента.
1 TACGGCTACC TTGTTACGAC TTACCCCAGT CGCTGACCCT ACCGTGGTCG AGCCCTCCCT
61 TGCGGGTTAG CGCATCGCCT TCGGGTAGAA CCAACTCCCA TGGTGTGACG GGCGGTGTGT
121 ACAAGGCCCG GGAACGTATT CACCGCGGCA TGCTGATCCG CGATTACTAG CAATTCCAAC
181 TTCATGCCCT CGAGTTGCAG AGGACAATCC GAACTGAGAC GACTTTTAAG GATTAACCCT
241 CTGTAGTCGC CATTGTAGCA CGTGTGTAGC CCACCCTGTA AGGGCCATGA GGACTTGACG
301 TCATCCCCAC CTTCCTCCGG CTTAGCACCG GCAGTCCCAT TAGAGTTCCC AACTAAATGA
361 TGGCAACTAA TGGCGAGGGT TGCGCTCGTT GCGGGACTTA ACCCAACATC TCACGACACG
421 AGCTGACGAC AGCCATGCAG CACCTGTGTC CTAGTCCCCG AAGGGAAAAC TGCAGATCTC
481 TCTGGCGGTC CAGGCATGTC AAAAGGTGGT AAGGTTCTGC GCGTTGCTGG AATTAAACCA
541 CATGCTCCAC CGCTTGTGCG GGCCCCGTCA ATTCCTTTCA GTTTTAATCT TGCGACCGTA
601 CTCCCCAGGC GGATTGCTTA ATGCGTTAGC TGCGTCACCG AGATGTAAAC ATCCCGACAA
661 CTAGCAATCA TGGTTTACGG CGTGAGCTAC CAGGGTATCT AATCCTGTTT GTTCCCCACG
721 CTTTCGAGCC TCAGCGTCAG TAATGATCCA GTATGTCGCC TTCGCCACTG GTGTTCTTCC
781 GAATATCTAC AATTCACCTC TACACTCGGA GTTCCACATA CCTCTATCAC ACTCAAGACA
841 CCCAGTATCA AAGGCAGTTC CAGAGTTGAG CTCTGGGATT TCACCCCTGA CTTAAATGTC
901 CGCCTACGCT CCCTTTAGCC AGTAATTCCG AGCAACGCTA GCCCCCTTCG TATTACCGCG
961 GCTGCTGGCA CGAAGTTAGC CGGGGCTTCT TCTGTAGGTA CCGTCATTAT CGTCCCTACT
1021 GAAAGAATTT TCAATCCTGA GGAGAAGCCT TCATCATTCA CGCGGCATGG CTGCGTCAGG
1081 CTTTCGCCCA TTGCGCAAGA TTCCCCACTG CTGCCTCCCG TAGGAGTTTG GGCCGTGTCT
1141 CAGTCCCAAT GTGGCTGGTC ATCCTCTCAG ACCAGCTACT GATCGTAGCC TTGGTGAGCC
1201 TTTACCTCAC CAACTAGCTA ATCAGACGCG GGCCGCTCTA AAGGTGATAA TCTTTTCCCC
1261 AAAGGGCACA TTCGGCATTA CCACCCGTTT CCAGGAGCTA TTCCGAGAAC CTAAAGGCAC
1321 GTTCCCACGT GTTACTCACC CGTCCGCCAC TAACCCCGAA GGGTCCGTTC GACTTGCATG
1381 TGTTAGGCCT GCCGCCAGCG TTCGCTCTGA GCCAGGATCA AACTC
На основе установленной последовательности фрагмента гена 16S рРНК определили принадлежность штамма-продуцента к Brevundimonas mongoliensis семейства Caulobacteraceae. Для этого использовали Nucleotide BLAST [6], определитель SILVAngs [7] и классификатор [8]. Морфология штамма соответствовала его виду [9]. Штамм назвали Brevundimonas mongoliensis 53, а выделенную из него рестриктазу, по номенклатуре, BmoI.
Получение препарата фермента BmoI и изучение его свойств.
В результате наработки культуры клеток штамма B. mongoliensis 53 из 7 л питательной среды получили 25 г биомассы. Активность целевого фермента в биомассе составила 2800 ед./г. После трёх стадий хроматографической очистки из 10 г биомассы было получено 4 мл препарата эндонуклеазы рестрикции BmoI с концентрацией 2000 е.а./мл.
В ходе дальнейшей работы нами были определены оптимальные условия для работы фермента. Для этого использовали реакционную смесь объемом 50 мкл, содержащую 1 мкг ДНК фага T7, один из шести буферов для эндонуклеаз рестрикции и 0,5, 1 или 2 мкл препарата фермента BmoI, разбавленного в 2 раза SE-буфером «B100» (10 мМ Трис-HCl, (pH 7,6), 50 мМ KCl, 0,1 мМ ЭДТА, 200 мкг/мл BSA, 1 мМ ДТТ, 50% глицерин). Смесь инкубировали при температуре 37oС в течение 1 ч. Результаты приведены на рисунке 2.
Рисунок 2
Активность препарата BmoI на ДНК фага T7 при температуре 37оС в разных SE-буферах: B, G, O, W, Y и ROSE. Электрофорез в 1% агарозном геле.
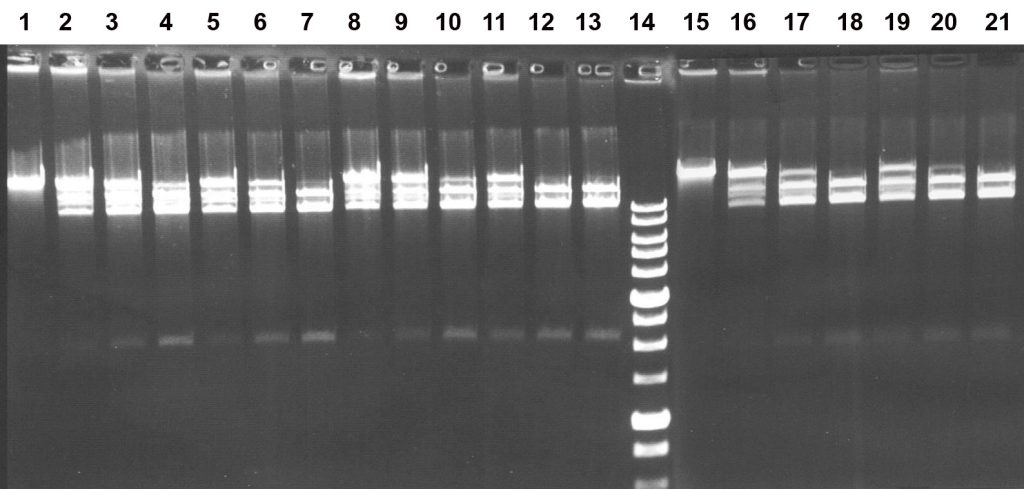
Дорожки:
1 и 15 – исходная ДНК фага T7;
2,5,8,11,16,19 – T7 ДНК + 0,5 мкл разбавленного в 2 раза BmoI;
3,6,9,12,17,20 – T7 ДНК + 1 мкл разбавленного в 2 раза BmoI;
4,7,10,13,18,21 – T7 ДНК + 2 мкл разбавленного в 2 раза BmoI;
2-4 – SE-буфер B;
5-7 – SE-буфер G;
8-10 – SE-буфер O;
11-13 – SE-буфер W;
16-18 – SE-буфер Y;
19-21 – SE-буфер ROSE;
14 – SE-маркёр молекулярных весов ДНК 1 kb.
Из рисунка 2 видно, что фермент проявляет максимальную активность в SE-буфере W (10 мМ Трис-HCl (pH 8,5), 10 мМ MgCl2, 100 мМ NaCl, 1 мМ ДТТ), поскольку только в нем наблюдается полный гидролиз субстратной ДНК с образованием трёх фрагментов при добавлении как 1, так и 2 мкл разбавленного в 2 раза фермента (дор.12-13). Во всех остальных случаях 1 мкл разбавленного препарата BmoI недостаточно для исчерпывающего гидролиза ДНК. Полученные результаты анализа активности фермента в шести SE-буферах при температуре 37оС представлены в таблице 1.
Таблица 1
Активность BmoI при 37°С в стандартных SE-буферах.
| B | G | O | W | Y | ROSE |
| 25–50 | 50–75 | 25–50 | 100 | 50–75 | 75 |
Сравнение полученной с помощью BmoI картины гидролиза ДНК фага T7 с теоретически рассчитанными данными расщепления ее последовательности по сайтам эндонуклеаз рестрикции позволило сделать вывод, что фермент BmoI является изошизомером рестриктазы SanDI, которая гидролизует данный субстрат по сайту 5’-GGGWCCC-3’ с образованием трёх фрагментов длиной 26794, 10989 и 2198 п.н. На ДНК фага λ сайты 5’-GGGWCCC-3’ отсутствуют, поэтому мы не наблюдаем гидролиза этого субстрата после его обработки ЭР BmoI (Рис. 3).
Рисунок 3
Определение специфичности эндонуклеазы рестрикции BmoI на ДНК фага λ и ДНК фага T7. Продукты разделены в 1% агарозном геле.
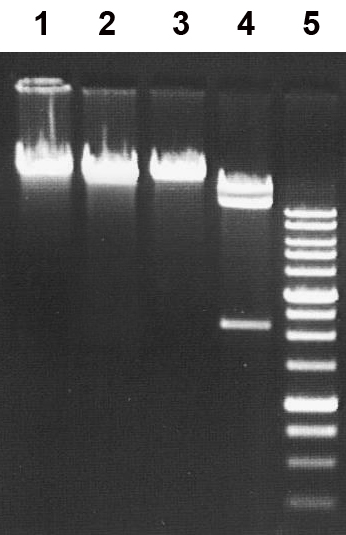
Дорожки:
1 – исходная ДНК фага λ;
2 – ДНК фага λ + 1 мкл препарата BmoI;
3 – исходная ДНК фага T7;
4 – ДНК фага T7 + 1 мкл препарата BmoI;
5 – SE-маркёр молекулярных весов ДНК 1 kb.
При определении оптимальной температуры для работы ЭР BmoI проводили инкубацию в 50 мкл реакционной смеси 1 мкг ДНК фага T7 в SE-буфере W и 0,5 или 1 мкл препарата фермента при различных температурах: 25, 30, 37, 45 или 55°С. Результаты представлены на рисунке 4.
Рисунок 4
Активность препарата BmoI на ДНК фага T7 в SE-буфере W при различных температурах. Электрофорез в 1% агарозном геле.
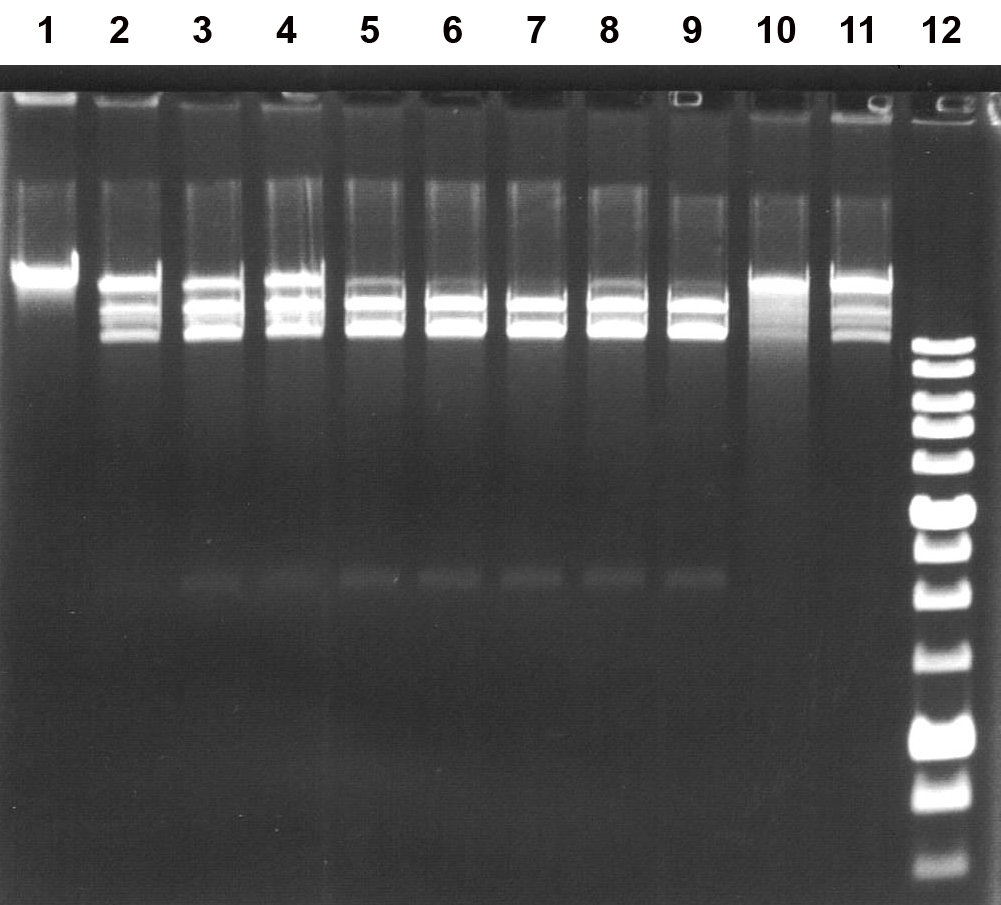
Дорожки:
1 – исходная ДНК фага T7;
2,3 – ДНК фага T7 + 0,5 и 1 мкл BmoI, 25оС;
4,5 – ДНК фага T7 + 0,5 и 1 мкл BmoI, 30оС;
6,7 – ДНК фага T7 + 0,5 и 1 мкл BmoI, 37оС;
8,9 – ДНК фага T7 + 0,5 и 1 мкл BmoI, 45оС;
10,11 – ДНК фага T7 + 0,5 и 1 мкл BmoI, 55оС;
12 – SE-маркёр молекулярных весов ДНК 1 kb.
Как видно из рис.4, полный гидролиз субстратной ДНК наблюдается при температуре 37оС (дор. 7), которая является оптимальной для активности рестриктазы BmoI. В то же время полная инактивация ЭР BmoI в реакционной смеси происходила при нагревании до 65oС за 20 мин (данные не приведены).
Из результатов, представленных на рисунках 2, 3 и 4, следует, что за единицу активности рестриктазы BmoI можно принять минимальное количество фермента, которое необходимо для полного расщепления 1 мкг ДНК фага T7 в реакционной смеси объемом 50 мкл за 1 ч в SE-буфере W при 37°С.
На рисунке 5 приведена электрофореграмма анализа активности целевого фермента при оптимальной температуре (37°С) в оптимальном SE-буфере W.
Рисунок 5
Определение активности BmoI в оптимальных условиях. Пробы по 50 мкл, содержащие 1 мкг ДНК фага T7 в SE-буфере W и 1 мкл неразбавленного или 2, 1, 0,5 мкл разбавленного в 2 раза буфером «B100» препарата BmoI. Смесь инкубировали 1 ч при 37°С и анализировали электрофорезом в 1% агарозном геле.
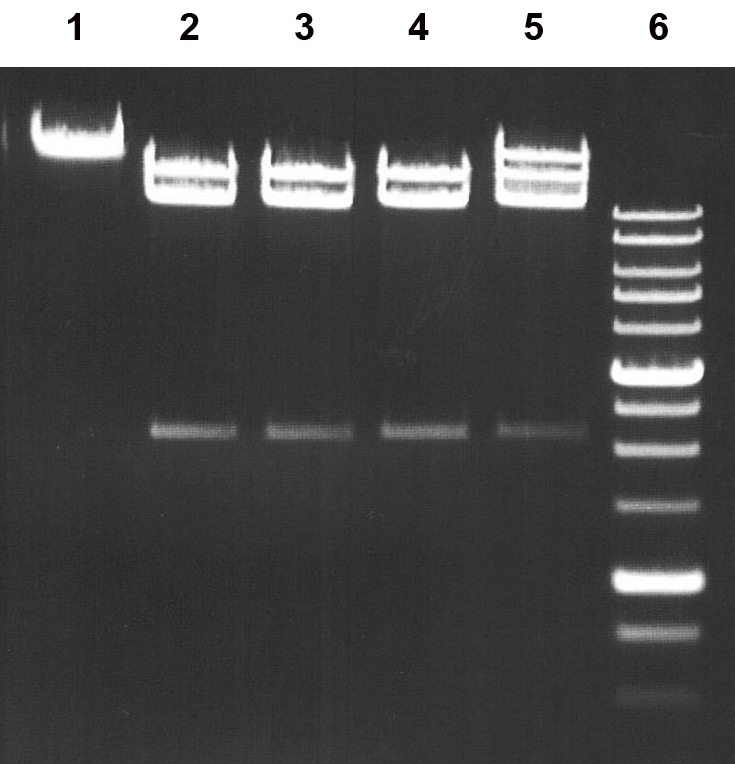
Дорожки:
1 – исходная ДНК фага T7;
2 – ДНК фага T7 + 1 мкл неразбавленного препарата BmoI;
3, 4, 5 – ДНК фага T7 + 2, 1 или 0,5 мкл разбавленного в 2 раза препарата BmoI, соответственно;
6 – SE-маркёр молекулярных весов ДНК 1 kb.
Поскольку мы наблюдаем исчерпывающий гидролиз ферментом Т7 ДНК на дорожках 2, 3 и 4, можно сделать заключение, что активность полученного препарата ЭР BmoI составляет 2 е.а./мкл (2000 е.а./мл).
Чтобы определить место гидролиза ДНК новым ферментом и подтвердить его специфичность мы использовали двуцепочечный радиоактивно-меченый с 5’-конца олигонуклеотидный дуплекс Bmo_SfaN*/ Bmo_SfaNk (меченая цепь отмечена *), содержащий сайт узнавания BmoI (выделен жирным и подчеркнут). Данный дуплекс содержит также сайты узнавания контрольных эндонуклеаз рестрикции SfaNI (5’-GCATC-3’(5/9), AspS9I (5’-G↓GNCC-3’) и BstF5I (5’-GGATG(2/0)-3’), расщепляющих его в позициях, указанных ниже стрелками:
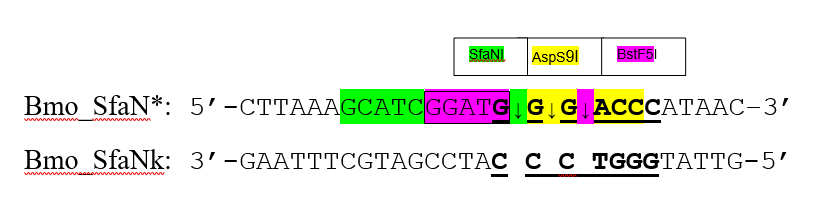
Меченый дуплекс расщепляли новым ферментом BmoI и отдельно контрольными ферментами SfaNI, AspS9I и BstF5I. Реакцию проводили с 1 пмоль дуплекса Bmo_SfaN*/ Bmo_SfaNk в 10 мкл реакционной смеси в течение 1 ч при 37°С в реакционном SE-буфере Y с добавлением 1 мкл одного из ферментов. Продукты гидролиза (по 10 мкл) наносили на 20% ПААГ с 8М мочевиной и проводили электрофорез в Трис-боратном буфере. На рисунке 6 представлена авторадиограмма геля после электрофореза.
Рисунок 6
Определение позиции гидролиза BmoI. Электрофорез в 20% ПААГ с 8М мочевиной.
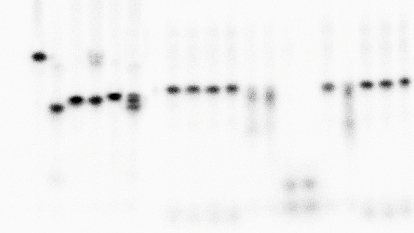
Дорожки:
1 – исходный дуплекс Bmo_SfaN*/ Bmo_SfaNk;
2 – дуплекс Bmo_SfaN*/ Bmo_SfaNk + 1 мкл SfaNI;
3 – дуплекс Bmo_SfaN*/ Bmo_SfaNk + 1 мкл AspS9I;
4 – дуплекс Bmo_SfaN*/ Bmo_SfaNk + 1 мкл BmoI;
5 – дуплекс Bmo_SfaN*/ Bmo_SfaNk + 1 мкл BstF5I;
6 – смесь из 3-х реакционных смесей, нанесенных на дорожки 2, 3 и 5 (SfaNI, AspS9I и BstF5I), по 2 мкл каждой.
Из данных, представленных на рисунке 6, следует, что позиция гидролиза BmoI совпадает с позицией гидролиза AspS9I (5’-G↓GNCC-3’). Следовательно, можно сделать вывод о том, что BmoI расщепляет последовательность узнавания между вторым и третьим гуанином симметрично на обеих цепях: 5’-GG↓GWCCC-3’/3’-CCCWG↓GG-5’.
Таким образом, фермент BmoI из штамма бактерии Brevundimonas mongoliensis 53 узнает семинуклеотидную палиндромную последовательность ДНК 5’-GG↓GWCCC-3’ и расщепляет ее после второго гуанина, образуя, таким образом трехнуклеотидные 5’-выступающие «липкие» концы. BmoI является изошизомером ранее описанной эндонуклеазы рестрикции SanDI и может применяться для исследований в области генетической инженерии, молекулярной биологии, биотехнологии и ДНК-диагностики.
Список литературы
- Timothy G. Simcox, Linda Fabian, Keith Kretz, Valerie Hedden, Mary Ellen C. Simcox. SanDI, a new type-II restriction endonuclease that recognizes 5′-GGGWCCC-3′ // Gene. – 1995. – V. 155 (1). – P. 129–130. DOI: 10.1016/0378-1119(94)00711-Z.
- Руководство к практическим занятиям по микробиологии. Под ред. Н.С. Егорова. М., 1995.
- Определитель бактерий Берджи. Под ред. Дж. Хоулта и др.: 9-е издание в 2-х томах. Перевод с англ. под ред. Г.А. Заварзина. М., 1997.
- Дедков В.С. Определение содержания G+C в ДНК бактерий при помощи эндонуклеаз рестрикции // Биотехнология. – 2004. – № 4. – С. 77–82.
- Weisburg W.G., Barns S.M., Pelletier D.A., Lane D.J. 16S ribosomal DNA amplification for phylogenetic study // J. Bacteriol. – 1991. – Vol. 173. – P. 697–703. DOI: 10.1128/jb.173.2.697-703.1991.
- Nucleotide BLAST. URL:https://blast.ncbi.nlm.nih.gov.
- SILVAngs. URL:https://www.arb-silva.de/aligner.
