Эндонуклеаза рестрикции Pse31I из штамма бактерии Peribacillus species 31, изошизомер Eco31I, узнает непалиндромную последовательность ДНК 5’-GGTCTC-3’ (1/5)
В.А. Чернухин1, В.С. Дедков1, Н.А. Михненкова1, Н.А. Воронцова1, М.А. Абдурашитов1, Д.А. Гончар1, С.Х. Дегтярев1
1 ОOO «СибЭнзайм» Новосибирск
* автор для переписки: Валерий Чернухин, ООО СибЭнзайм, ул. Тимакова, д. 2/12, Новосибирск, 630117, Россия; тел.: +7(383)333-4991; факс: +7(383)333-6853; E-mail: valerunishe@mail.ru
Нами обнаружен бактериальный штамм Peribacillus species 31, который является продуцентом новой эндонуклеазы рестрикции, названной Pse31I. Фермент узнает шестинуклеотидную непалиндромную последовательность ДНК 5’-GGTCTC-3’и расщепляет ее в стороне от сайта узнавания в позициях 1/5: 5’-GGTCTC(N)1^/3’-CCAGAG(N)5^-5’. Таким образом, Pse31I является истинным изошизомером рестриктазы Eco31I [1]. Штамм-продуцент идентифицировали по морфологическим и биохимическим признакам, а также по анализу первичной структуры фрагмента гена 16S рРНК. Pse31I имеет максимальную активность при 37°С и инактивируется инкубацией при 55 и 65°С. Фермент не расщепляет сайты с метилированным цитозином в верхней цепи (5’-GGTCT(5mC)-3’) и/или в нижней цепи (3’-(5mС)CAGAG).
Оптимальными условиями работы фермента являются SE-буфер 5 (Y) (33 мМ Трис-ацетат (pH 7,9), 10 мМ Mg-ацетат, 66 мМ K-ацетат, 1 мМ ДТТ) и температура 37°С.
Препарат эндонуклеазы рестрикции PecI с концентрацией 10000 ед/мл был получен путем очистки в четыре хроматографические стадии. Оптимальными условиями реакции фермента являются SE-буфер 5 (Y) (33 мМ Трис-ацетат (pH 7.9), 10 мМ Mg-ацетат, 66 мМ K-ацетат, 1 mM ДТТ) и температура 37oС.
Список сокращений: БСА – бычий сывороточный альбумин; ДТТ – дитиотреитол; Т7 ДНК – ДНК бактериофага T7; λ ДНК – ДНК бактериофага λ; Трис – трис-(оксиметил)аминометан; ПААГ – полиакриламидный гель; рестриктаза, ЭР – эндонуклеаза рестрикции; ЭДТА – этилендиамин-тетрауксусная кислота; SАМ – S-аденозил-L-метионин; M.SssI – ДНК-метилтрансфераза SssI; (5mC) — 5-метилцитозин;
Ключевые слова: штамм-продуцент, выделение фермента, эндонуклеаза рестрикции IIS-типа, чувствительность к (5mC)G-метилированию ДНК
DOI: 10.26213/3034-4301.2025.4.1.001
Данные для цитирования:
В.А. Чернухин, В.С. Дедков, Н.А. Михненкова, Н.А. Воронцова, М.А. Абдурашитов, Д.А. Гончар, С.Х. Дегтярёв. (2025) Эндонуклеаза рестрикции Pse31I из штамма бактерии Peribacillus species 31, изошизомер Eco31I, узнает непалиндромную последовательность ДНК 5’-GGTCTC-3’ (1/5), ДНК-узнающие ферменты, том 2025(1), DOI: 10.26213/3034-4301.2025.4.1.001

Эта статья доступна по лицензии Creative Commons Attribution-NonCommercial-NoDerivatives 4.0 International
ВВЕДЕНИЕ.
Рестриктазы IIS-типа [2] расщепляют ДНК вне сайта узнавания, образуя уникальные липкие концы, что позволяет их использовать в некоторых технологиях генетической инженерии. В данной работе изучен один из таких ферментов Pse31I.
УСЛОВИЯ ЭКСПЕРИМЕНТА
Штамм Peribacillus sp. 31 выращивали в LM-бульоне, содержащем 1% триптон (Organotechnie, Франция), 0,5% дрожжевой экстракт (той же фирмы), 0,5% NaCl, 0,05% MgCl2 и 0,001% тиамин. Рассевали на LM-агаре – 1,5% агар (C.E. Roeper Gmbh, Германия) в LM-бульоне. Среды готовили по 0,3 л в 0,5-литровых конических колбах. Для получения биомассы культуру выращивали в колбах с бульоном при 25оС со встряхиванием 2 дня. Клетки собирали центрифугированием на центрифуге Beckman J2-MI (США) в роторе JA-10 при 4оС, 8000 об/мин в течение 20 мин. С 10 л получали 43,7 г биомассы, которую замораживали при -20оС.
Очистка препарата эндонуклеазы рестрикции Pse31I
Условия проведения выделения и используемые буферы. Выделение проводили при +4°С с использованием буферных растворов:
буфер А — 10 мМ Трис-HCl, pH 7,6; 0,1 мМ ЭДТА и 7 мМ β-меркаптоэтанол;
буфер Б — 60 мМ Трис-HCl, pH 7,6; 0,1 мМ ЭДТА и 7 мМ β-меркаптоэтанол, 1 М сульфат аммония;
Экстрагирование. 43,7 г биомассы суспендировали в 150 мл буфера А, содержащего 0,1% Тритон Х–100, 0,2 M NaCl, 1 мг/мл лизоцим, 0,1 мМ PMSF (фенилметил-сульфонилфторид – ингибитор протеаз) в течение 2 часов на магнитной мешалке. Далее суспензию клеток в стеклянном стакане на 200 мл термостатировали в ледяной бане, затем клетки разрушали ультразвуком при максимальной мощности с амплитудой 22–24 мкм на приборе Soniprep 150 («MSE», Англия) с насадкой диаметром 2 см десятью импульсами по 2 мин с интервалом 2 мин. Экстракт осветляли на центрифуге J2-М1 («Beckman», США) в течение 30 минут в роторе JA-20 при 15000 об/мин. Осадок отбрасывали.
Хроматография на фосфоцеллюлозе Р-11. Супернатант смешивали со 100 мл фосфоцеллюлозы, предварительно промытой 500 мл буфера А, содержащего 0,1 М NaCl, 0,01% Тритон X-100 в стакане в течение 30 мин. После инкубации смесь переносили в колонку, промывали 100 мл буфера А, фермент элюировали линейным градиентом концентрации NaCl 0,1–0,5 М с 0,01% Тритоном X-100 в буфере А, объемом 1000 мл. Собирали 100 фракций по 10 мл. Объединяли фракции, содержащие максимум целевой активности, куда добавляли Трис-HCl, pH 7,6, до концентрации 60 мМ и сульфат аммония до концентрации 1 М, после чего фермент наносили на колонку с фенил-сефарозой объемом 5 мл, уравновешенную буфером Б.
Хроматография на фенил-сефарозе. Сорбированный материал элюировали линейным градиентом концентрации (NH4)2SO4 от 1 М до 0 М общим объемом 300 мл. Собирали 50 фракций по 6 мл. Объединяли фракции, содержащие максимум активности, к которым добавили 300 мкл БСА (10 мг/мл). Полученный раствор с целевой активностью диализовали против 1500 мл буфера А, содержащего 1% глицерин, в течение ночи. Утром ставили повторный диализ в 900 мл буфера А, содержащего 0,1 М NaCl и 1% глицерин в течение 1 часа.
Хроматография на гепарин-сефарозе. Диализованный фермент наносили на колонку с 5 мл гепарин-сефарозы, уравновешенной буфером А с 0,1 М NaCl. Элюировали фермент буфером А с линейным градиентом концентрации NaCl от 0,1 М до 0,5 М объемом 100 мл. Получили 50 фракций по 2 мл.
Активные фракции концентрировали в буфере А с 0,2 М NaCl и 55% глицерином.
В результате получили 2 мл целевого препарата Pse31I с активностью 10000 ед./мл.
Активность рестриктазы определяли на ДНК бактериофага T7 (ООО «СибЭнзайм», Россия). Использовали SE-буферы производства «СибЭнзайм»: буфер B – 10 мМ Трис-HCl (pH 7,6), 10 мМ MgCl2, 1 мМ ДТТ; буфер G – 10 мМ Трис-HCl (pH 7,6), 10 мМ MgCl2, 50 мМ NaCl, 1 мМ ДТТ; буфер O – 50 мМ Трис-HCl (pH 7,6), 10 мМ MgCl2, 100 мМ NaCl, 1 мМ ДТТ; буфер W – 10 мМ Трис-HCl (pH 8,5), 10 мМ MgCl2, 100 мМ NaCl, 1 мМ ДТТ; буфер Y – 33 мМ Трис-ацетат (pH 7,9), 10 мМ магний-ацетат, 66 мМ калий-ацетат, 1 мМ ДТТ; буфер ROSE – 17 мM Трис-ацетат pH 7,9, 5 мM Трис-HCl pH 7,9, 5 мM магний-ацетат, 5 мM MgCl2, 33 мM калий-ацетат, 50 мМ NaCl, 1 мM ДТТ. В качестве ДНК-маркера использовали гидролизат λ ДНК/BssT1I с фрагментами: 19329, 7743, 6223, 4254, 3472, 2690, 1882, 1489, 925, 421 и 74 п.н. того же производителя, в концентрации 50 мкг/мл, который наносили на гель для электрофореза по 0,3 мкг.
Расщепление ДНК проводили в пробах по 50 мкл, содержащих 50 мкг/мл Т7 ДНК, 100 мкг/мл БСА и SE-буфер. Для электрофореза в пробы добавляли по 10 мкл Стоп-буфера (100 мМ Na-ЭДТА (pH 8,5), 50% сахарозы и 0,3% Понсо 4R (Roha, Индия). Электрофорез проводили в 0,8% агарозе (Conda, Испания) в буфере ТАЕ (40 мМ Трис-ацетат, (pH 8,0), 20 мМ Na-ацетат, 1 мМ Na-ЭДТА с 0,5 мг/л бромистого этидия) при 5 В/см в течение 1 ч.
Гель фотографировали в ультрафиолете через оранжевый светофильтр на установке Herolab (Германия).
За единицу активности рестриктазы принимали минимальное количество фермента, необходимое для полного расщепления 1 мкг Т7 ДНК за 1 ч в SE-буфере Y при 37оС.
Для получения 5mCG-метилированной ДНК смесь объемом 1 мл, содержащую 500 мкг/мл Т7 ДНК в 10 мМ Трис-HCl (pH 7,6), 1 мМ ЭДТА; 0,1 мл 10-кратного буфера О с добавлением 20 мкл 0,5 М SАМ (Гептрал®, Фамар Л’Эйль, Франция) в 0,2 М H2SO4 с 20 мкл 2,5 М Трис-основного; 20 мкл БСА и 50 мкл M.SssI с активностью 1 ед/мкл («СибЭнзайм») инкубировали при 24оС в течение 20 ч, прогревали при 65оС 20 мин и хранили при -18оС.
РЕЗУЛЬТАТЫ И ОБСУЖДЕНИЕ
Характеристика и таксономическая идентификация штамма. Штамм выделен из образца почвы. На LM-агаре за 3 дня при 24оС образует круглые колонии до 5 мм в диаметре. Колонии выпуклые, зернисто-блестящие, непрозрачные, серые. Клетки палочковидные, 0,7 х 5 – 20 мкм, неподвижные, грамположительные, каталазоположительные, оксидазоотрицательные, аэробные, образуют споры. Растут при температуре от 8 до 37оС и не растут при 55оС.
Для идентификации штамма по 16S рРНК из клеток штамма выделяли ДНК и проводили ПЦР гена 16S рРНК с использованием праймеров 5’-agagtttgatcmtggctca-3’ и 5’-tacggytaccttgttacgact-3’, представляющих модификации fDl и rPl [3]. Секвенировали 1440 нуклеотидов части гена 16S рРНК (рис. 1).
Рисунок 1
Нуклеотидная последовательность фрагмента гена 16S рРНК (длина 1440 п.н.) штамма-продуцента.
На основе установленной последовательности фрагмента гена 16S рРНК определили принадлежность штамма-продуцента к роду Peribacillus семейства Bacillaceae, используя Nucleotide BLAST [4] и определитель SILVAngs [5]. Морфология штамма также соответствовала его семейству [6]. Штамм назвали Peribacillus species 31, а выделенную из него рестриктазу, по номенклатуре, Pse31I.
Определение оптимального SE-буфера и температуры работы фермента проводили путём инкубации проб по 50 мкл, содержащих 50 мкг/мл Т7 ДНК, 100 мкг/мл БСА, SE-буфер: B, G, O, W, Y или R (ROSE) – и 1% по объёму препарата Pse31I при 24 оС, 37 оС и 55оС в течение 1 ч с последующим электрофорезом в 0,8% агарозном геле. Полученная электрофореграмма представлена на рисунке 2.
Как видно из этого рисунка рестриктаза Pse31I наиболее активна в SE-буферах Y и R при 37оС (рис. 2, таблица 1). При 55оС активность незначительна, вероятно, из-за инактивации фермента.
Рисунок 2
Активность Pse31I в расщеплении Т7 ДНК при разной температуре в SE-буферах: B, G, O, W, Y и R (ROSE); М – маркер λ/BssT1I. Пробы, содержащие 50 мкг/мл Т7 ДНК, 100 мкг/мл БСА, SE-буфер и 1% по объёму Pse31I, инкубировали 1 ч и анализировали электрофорезом в 0,8% агарозе.
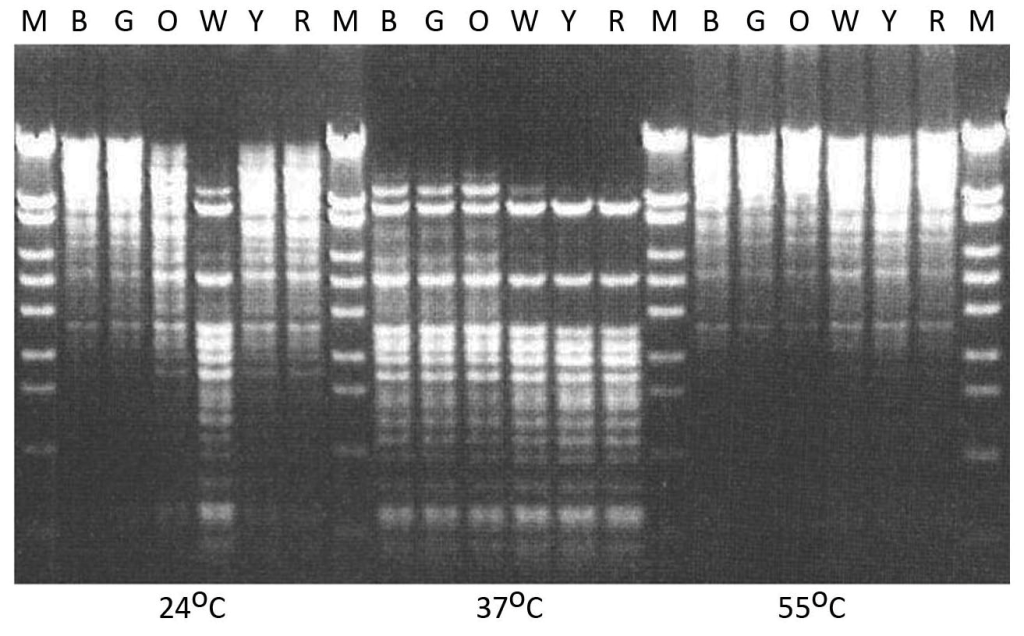
Таблица 1
Активность Pse31I при 37оС в стандартных SE-буферах
| B | G | O | W | Y | ROSE |
| 25-50 | 75-100 | 10-25 | 25-50 | 100 | 75 |
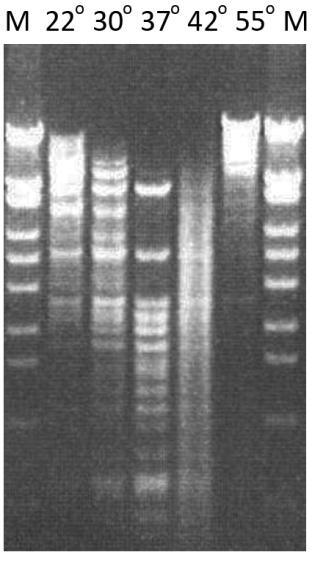
На рисунке 3 приведен анализ активности целевого фермента при разных температурах. Как видно из этого рисунка, рестриктаза в оптимальном SE-буфере Y наиболее активна при 37оС, а при 42 оС наблюдается дополнительная нуклеазная активность
Рисунок 3
Активность Pse31I в расщеплении Т7 ДНК при разной температуре в SE-буфере Y. М – маркер λ/BssT1I. Пробы по 50 мкл, содержащие 50 мкг/мл Т7 ДНК, 100 мкг/мл БСА, SE-буфер и 1% по объёму Pse31I, инкубировали 1 ч и анализировали электрофорезом в 0,8% агарозе
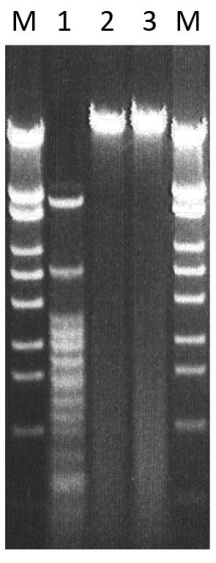
Как оказалось, Pse31I инактивируется прогреванием при 55оС и 65оС за 20 мин, что следует из результатов, представленных на рисунке 4.
Рисунок 4
Активность Pse31I после прединкубации 1 мкл фермента в SE-буфере Y с 50 мкг/мл ДНК фага Т7М в реакционной смеси объемом 50 мкл при различных температурах: 1 — при 24оС 20 мин; 2 — при 55оС 20 мин; 3 — при 65оС 20 мин; после прединкубации проводили гидролиз ДНК 1 ч при 37оС. Маркер — λ/BssT1I. Электрофорез в 0,8% агарозе
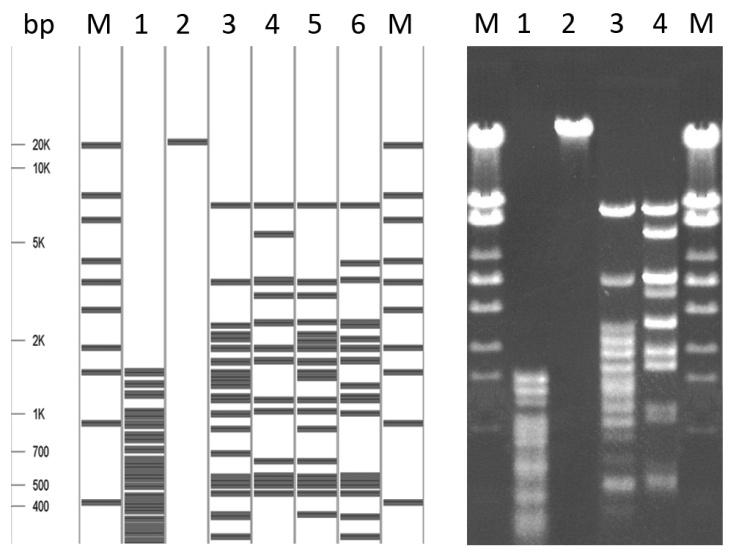
Чувствительность Pse31I к (5mC)G-метилированию определяли на ДНК фага Т7, метилированной M.SssI, как описано в условиях эксперимента. Контролем метилирования является устойчивость модифицированной ДНК к расщеплению ЭР HspAI (G^CGC) (дорожки 1 и 2). Pse31I узнаёт непалиндромный сайт GGTCTC, крайние цитозины которого могут быть метилированы M.SssI по одной из цепей ДНК: GGTCTCg и GAGACCg, реже, по обеим. Моделирование метилирования по [7] показало, что на Т7 ДНК можно выявить (5mC)G-метилирование каждой цепи сайта, узнаваемого Pse31I по блокированию его расщепления (рис. 5, дорожки 3,4,5 и 6). В эксперименте метилирование как цепи с сайтом GGTCT(m5C), так и цепи с сайтом GAGAC(m5C) блокировало Pse31I на 95–100% (рис. 5, дорожки 3 и 4).
Рисунок 5
Расчётное и экспериментальное определение чувствительности Pse31I к 5mCG-метилированию Т7 ДНК: 1 – исходная ДНК + HspAI (G^CGC), 2 – (5mC)G-метилированная ДНК + HspAI, 3 – исходная ДНК + Pse31I (GGTCTC(1/5), 4 – (5mC)G-метилированная ДНК + Pse31I, 5 – GGTCT(m5C)-метилированная ДНК + Pse31I, 6 – GAGAC(m5C)-метилированная ДНК + Pse31I, М – λ ДНК + BssT1I. 50 мкг/мл Т7 ДНК в SE-буфере Y инкубировали со 100 ед/мл Pse31I при 37оС в течение 1ч. Электрофорез в 0,8% агарозе
Определение позиции гидролиза ДНК ферментом. Чтобы определить место гидролиза ДНК новым ферментом мы использовали радиоактивно-меченый с 5’-конца олигонуклеотидный дуплекс Bso31I*/ Bso31IК (меченая цепь отмечена «*»), содержащий сайт узнавания Bso31I (подчеркнут):
Bso31I*: 5’-AGCTTCCTAGGTCTCCGGAATATTAATGACTCGAGT-3’
Bso31IK: 5’-ACTCGAGTCATTAATATTCCGGAGACCTAGGA-3’
Данный дуплекс расщепляли новым ферментом Pse31I и отдельно контрольным ферментом Bso31I.
Реакцию проводили с 1 пмоль дуплексов Bso31I*/ Bso31IК и Bso31IK*/ Bso31I в 10 мкл реакционной смеси в течение 1 ч при 37°С в реакционном SE-буфере ”Y”. Продукты гидролиза наносили на 20% ПААГ с 8М мочевиной и проводили электрофорез в Трис-боратном буфере. На рисунке 6 представлена авторадиограмма геля после электрофореза.
Рисунок 6
Определение позиции гидролиза Pse31I на олигонуклеотидном дуплексе Bso31I/Bso31IК. Дорожки: 1 – исходный дуплекс Bso31I*/Bso31IК; 2 –дуплекс Bso31I*/Bso31IК + 1 мкл Pse31I; 3 – дуплекс Bso31I*/Bso31IК + 1 мкл Bso31I; 4 – исходный дуплекс Bso31IK*/Bso31I; 2 –дуплекс Bso31IK*/Bso31I + 1 мкл Pse31I; 3 – дуплекс Bso31IK*/Bso31I + 1 мкл Bso31I. Электрофорез в 20% ПААГ с 8М мочевиной
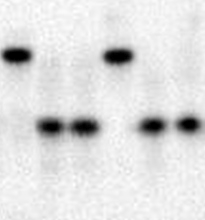
Из данных, представленных на рисунке 6, следует, что позиции гидролиза Pse31I совпадают с позициями гидролиза Bso31I: оба фермента расщепляют двуцепочечную ДНК в стороне от сайта узнавания 5’-GGTCTC-3’ в позициях 1/5. Таким образом, Pse31I образует в результате гидролиза ДНК четырёхнуклеотидный 5’-выступающий конец и является истинным изошизомером Bso31I.
Новый фермент Pse31I может найти применение в области генетической инженерии, молекулярной биологии, биотехнологии и ДНК-диагностики. Благодаря 100%-ной активности в универсальном SE-буфере “ROSE” и эффективном гидролизе ДНК при типичной для большинства эндонуклеаз рестрикции температуре 37°С, а также возможности инактивации фермента при 65°С, данную ЭР можно использовать в различных экспериментах.
Список литературы
- Butkus V., Bitinaite J., Kersulyte D., Janulaitis A. A new restriction endonuclease Eco31I recognizing a non-palindromic sequence. Biochim Biophys Acta. 1985 Dec 18;826(4):208-12. doi: 10.1016/0167-4781(85)90008-9.
- Pingouda A., Fuxreiterb M., Pingouda V. and Wendea W. Type II restriction endonucleases: structure and mechanism. CMLS, Cell. Mol. Life Sci. 62 (2005) 685–707. 1420-682X/05/060685-23. DOI 10.1007/s00018-004-4513-1
- Weisburg W.G., Barns S.M., Pelletier D.A., Lane D.J. 16S ribosomal DNA amplification for phylogenetic study // J. Bacteriol. – 1991. – Vol. 173. – P. 697 – 703., DOI: 10.1128/jb.173.2.697-703.1991
- 4. Nucleotide BLAST. URL: https://blast.ncbi.nlm.nih.gov
- 5. SILVAngs. URL: https://www.arb-silva.de/aligner
- URL: https://www.ncbi.nlm.nih.gov/Taxonomy/
- Дедков В.С. Определение специфичности ДНК-метилтрансферазы M.Bsc4I в клеточном лизате при помощи блокирования эндонуклеаз рестрикции и компьютерного моделирования // Молекул. генетика, микробиол. и вирусол. – 2009. – № 3. – С. 3-8.
